



Next: Abréviations
Up: Recherche de gènes de
Previous: List of Figures
Contents
- Exemples de quelques maladies multifactorielles où le facteur de risque génétique a été confirmé
- Fréquence de quelques maladies inflammatoires et auto-immunes dans la population générale et chez les germains de premier degré du probant ainsi que les valeurs du risque relatif (
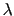 s)
s)
- Fréquence des SNPs présents dans la population humaine
- Nombre de SNPs présents à l'intérieur ou proches des régions codantes
- Rôle des cellules T effectrices dans les réponses de type cellulaire et de type humoral contre les pathogènes
- Hiérarchie des choix thérapeutiques du psoriasis vulgaire
- Motivations pour initier un traitement systémique du psoriasis
- Exemples de thérapies systémiques du psoriasis
- Taux de concordance de différentes maladies multifactorielles chez les jumeaux monozygotes et dizygotes
- Liste des loci de prédisposition au psoriasis et caractéristiques des populations utilisées pour les études
- Gènes et variants associés avec le psoriasis retrouvés dans au moins une étude
- Modèles murins et leur ressemblance au psoriasis humain
- Structure et caractéristiques généalogiques et cliniques des 126 familles du Lot I et du Lot II
- Microsatellites supplémentaires génotypés dans les 14 premières familles du Lot I ou dans la totalité des 46 familles du même lot
- Mélange réactionnel pour une amplification individuelle du marqueur par PCR sur une plaque de 96 puits
- Différentes étapes du cycle de la PCR le plus souvent utilisé
- Liste des amorces choisis pour chaque exon du HLA-C
- Conditions choisies pour l'amplification des exons 2 et 3 du gène HLA-C
- Mélange réactionnel pour une amplification individuelle des exons 2 et 3 par PCR sur une plaque de 96 puits
- Différentes étapes du cycle de la PCR, variant selon la température d'hybridation des amorces pour chaque exon
- Résumé des résultats les plus significatifs (Z>2) de l'analyse de liaison paramétrique réalisé sur la totalité des marqueurs (Criblage 1+2) sur les 46 familles françaises par MLINK
- Identification et analyse d'association des TagSNPs de PTPN22 sur 45 familles (Lot I) par FBAT
- Analyse d'association des haplotypes de PTPN22 sur le Lot I par FBAT
- Analyse d'association des TagSNPs de PTPN22 sur le Lot I avec la méthode "LNMs"
- Analyse d'association des TagSNPs de PTPN22, suggestifs dans le Lot I, faite sur un deuxième lot de 83 familles (Lot II) et sur l'ensemble des familles (Lot I et Lot II)
- Analyse d'association des TagSNPs de PTPN22 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et test d'association des TagSNPs de CARD15 sur le Lot I par FBAT
- Analyse d'association des haplotypes de CARD15 sur le Lot I par FBAT
- Analyse d'association des TagSNPs de CARD15 dans le Lot I avec la méthode "LNMs"
- Analyse d'association des TagSNPs de CARD15, suggestifs dans le Lot I, faite sur le Lot II et ainsi que sur l'ensemble des lots (Lot I et Lot II)
- Analyse d'association des TagSNPs de CARD15 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et analyse d'association des TagSNPs de CYLD sur le Lot I par FBAT
- Analyse d'association des haplotypes de CYLD sur le Lot I par FBAT
- Analyse d'association des TagSNPs de CYLD dans le Lot I avec la méthode "LNMs"
- Analyse d'association des TagSNPs de CYLD, suggestifs dans le Lot I, faite sur un deuxième lot de 83 familles (Lot II) et sur l'ensemble des lots (Lot I et Lot II)
- Analyse d'association des TagSNPs de CYLD dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et test d'association des TagSNPs de SLC9A3R1/NAT9 sur le Lot I par FBAT
- Analyse d'association des haplotypes de SLC9A3R1/NAT9 sur le Lot I par FBAT
- Analyse d'association des TagSNPs de SLC9A3R1/NAT9 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et test d'association des TagSNPs de RAPTOR sur le Lot I par FBAT
- Analyse d'association des haplotypes de RAPTOR sur le Lot I par FBAT
- Analyse d'association des TagSNPs de RAPTOR dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et test d'association des TagSNPs de SLC22A4 et SCL22A5 sur le Lot I par FBAT
- Analyse d'association des haplotypes de SLC22A4 et de SLC22A5 sur le Lot I par FBAT
- Analyse d'association des TagSNPs de SLC22A5 dans le Lot I avec la méthode "LNMs"
- Analyse d'association du seul TagSNP de SLC22A5, suggestif dans le Lot I, faite sur le Lot II et ainsi que sur l'ensemble des lots (Lot I et Lot II)
- Analyse d'association des TagSNPs de SLC22A4 et de SLC22A5 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et analyse d'association des TagSNPs de IL1R1, IL1A, IL1B et IL1RN sur le Lot I par FBAT
- Analyse d'association des haplotypes de IL1R1, IL1A, IL1B et IL1RN sur le Lot I par FBAT
- Analyse d'association des TagSNPs dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et analyse d'association des TagSNPs de IL12RB1 sur le Lot I par FBAT
- Analyse d'association des haplotypes de IL12RB1 sur le Lot I par FBAT
- Analyse d'association des TagSNPs de IL12RB1 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et analyse d'association des TagSNPs dans SLC12A8 sur le Lot I par FBAT
- Analyse d'association des haplotypes de SLC12A8 sur le lot I par FBAT
- Analyse d'association des TagSNPs de SLC12A8 dans le lot I avec la méthode "LNMs"
- Analyse d'association des TagSNPs de SLC12A8, suggestifs dans le lot I, faite sur le Lot II et ainsi que sur l'ensemble des lots (Lot I et Lot II)
- Analyse d'association des TagSNPs de SLC12A8 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et analyse d'association des TagSNPs de FLG sur le Lot I par FBAT
- Analyse d'association des TagSNPs de FLG dans le Lot I avec la méthode "LNMs"
- Analyse d'association des haplotypes de FLG sur le Lot I par FBAT
- Analyse d'association des TagSNPs de FLG, suggestifs dans le Lot I, faite sur le Lot II et ainsi que sur l'ensemble des lots (Lot I et Lot II)
- Analyse d'association des TagSNPs deFLG dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et analyse d'association des TagSNPs de TGM1, TGM3, TGM5 et TGM6 sur le Lot I par FBAT
- Analyse d'association des haplotypes de TGM1, TGM3, TGM5 et TGM6 sur le Lot I par FBAT
- Analyse d'association des TagSNPs de TGM1, TGM3, TGM5 et TGM6 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et analyse d'association des TagSNPs de STAT3 sur le Lot I par FBAT
- Analyse d'association des TagSNPs de STAT3 dans le Lot I avec la méthode "LNMs"
- Analyse d'association du TagSNP de STAT3, suggestif dans le Lot I, faite sur le Lot II et ainsi que sur l'ensemble des lots (Lot I et Lot II)
- Analyse d'association des haplotypes de STAT3 sur le Lot I par FBAT
- Analyse d'association des TagSNPs de STAT3 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
- Identification et test d'association des TagSNPs de JUN, JUNB sur le Lot I par FBAT
- Analyse d'association des haplotypes de JUN sur le Lot I par FBAT
- Analyse d'association des TagSNPs de JUN dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
anouar
2009-08-22