| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
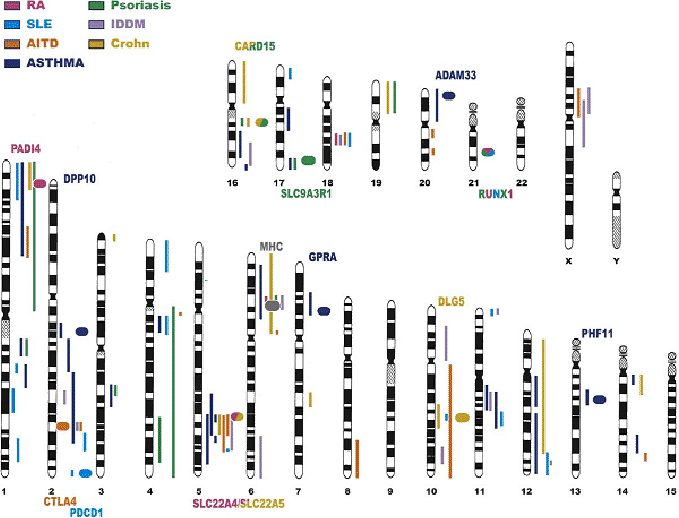 |
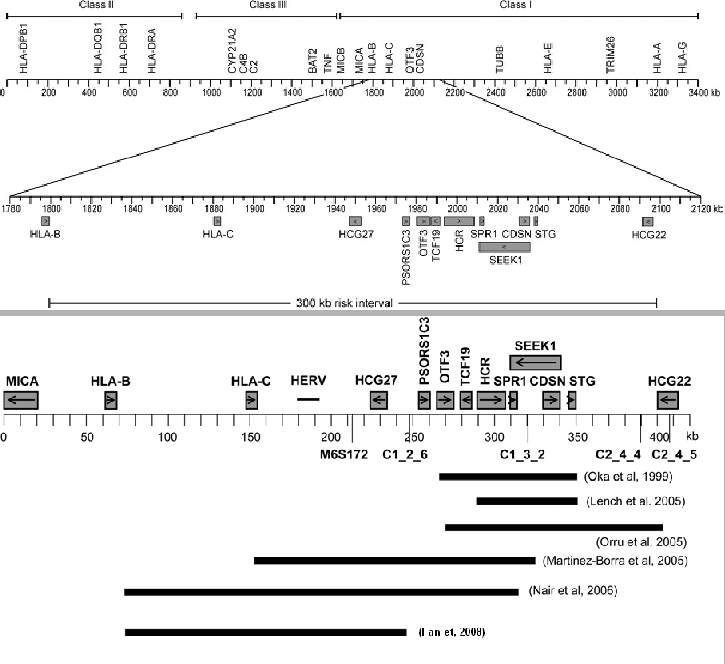
de classe I du système HLA (1.8Mb) sur le chromosome 6p21.3, Fig 1.14Cartographie de la région PSORS1 montrant la position des gènes connus ainsi que les régions candidates minimales pour contenir le gène causal identifiés lors des différentes études. En effet, lors de diverses études de liaison paramétriques et non paramétriques, ce locus d'environ 300kb a été identifié et confirmé par l'existence d'une forte liaison entre le psoriasis et plusieurs marqueurs présents dans la région dans différentes populations de diverses ethnies (américaine, européenne (allemande, suédoise, anglaise, islandaise), chinoise) (Tableau 1.10Liste des loci de prédisposition au psoriasis et caractéristiques des populations utilisées pour les études). Ce locus serait responsable d'un tiers à un demi de la part génétique du psoriasis [Trembath et al., 1997,Allen et al., 2003]. Des analyses d'associations sur cette région soutiennent également l'importance du locus PSORS1 dans la susceptibilité au psoriasis.
A l'aide de nombreux marqueurs espacés au niveau de la région HLA, plusieurs études d'association ont tenté de rétrécir ce large locus afin d'identifier le gène causatif [Balendran et al., 1999,Oka et al., 1999,Nair et al., 2000,Veal et al., 2002,Helms et al., 2005,Lench et al., 2005,Martinez-Borra et al., 2005,Orru et al., 2005,Nair et al., 2006,Fan et al., 2008](Figure 1.14Cartographie de la région PSORS1 montrant la position des gènes connus ainsi que les régions candidates minimales pour contenir le gène causal identifiés lors des différentes études).
Basé sur ces études, le gène de prédisposition au psoriasis du locus PSORS1 a été localisé dans une région d'environ 200kb du coté télomérique du HLA-C. Cette région contient une dizaine de gènes dont certains sont des candidats potentiels : HLA-C, TCF19 (SCI), OTF3 (POU5FI), HCR (Pg8), CDSN, SEEK1, SPR1, and STG (Figure 1.14Cartographie de la région PSORS1 montrant la position des gènes connus ainsi que les régions candidates minimales pour contenir le gène causal identifiés lors des différentes études). Deux d'entre eux, HCR (helix coiled coil rod homolog) et CDSN (corneodesmosin), furent activement étudiés pour leur rôle éventuel dans la pathogenèse de la maladie.
Le gène HCR a tout d'abord été prédit lors d'une étude sur la séquence génomique de la région HLA. Nommé tout d'abord Pg8 (putative gene 8) puis, plus récemment HCR, ce gène hautement polymorphe code pour une protéine alpha hélicoïdale de fonction inconnue [Guillaudeux et al., 1998,Oka et al., 1999,Asumalahti et al., 2000,Asumalahti et al., 2002]. Ce gène est situé dans l'une des régions candidates de PSORS1 identifiée lors de diverses études globales de la région dans les populations japonaises, américaines ou européennes [Oka et al., 1999,Nair et al., 2000,Martinez-Borra et al., 2005,Orru et al., 2005]. Une première étude d'association (cas-témoins) sur le gène lui-même, comparant 93 témoins avec 100 patients finlandais atteints de psoriasis démontre une association de ce gène avec le psoriasis [Asumalahti et al., 2000]. Deux SNPs, présents dans l'exon 4 du gène HCR (307 C>T et 325 C>T (Arginine en Tryptophane)) sont conjointement associés de manière significative au psoriasis (42% pour les individus atteints versus 20% pour les témoins, P= 0.00068) [Asumalahti et al., 2000]. Ces deux marqueurs, prédits pour changer la structure secondaire de la protéine, semblent être, en plus, en fort déséquilibre de liaison chez les patients et chez les témoins avec l'allèle HLA-Cw6, qui est aussi fortement associé avec la maladie (P=0.0000031) [Asumalahti et al., 2000,Asumalahti et al., 2002].
 |
Une autre étude cas-témoins, réalisée sur 419 familles venant de 6 populations (Grande Bretagne, Finlande, Suède, Espagne, Italie, Inde), confirme l'association du gène HCR avec le psoriasis, particulièrement avec un haplotype constitué de 4 SNPs codants non-synonymes (307T, 325T, 1723T et 2327G), HCR*WWCC (P=1x10![]() ). Les trois premiers variants semblent intervenir dans la structure secondaire de la protéine [Asumalahti et al., 2002]. Un ou l'ensemble de ces variants est montré(s) également associé(s) avec la maladie, et particulièrement avec le psoriasis de type I, dans d'autres populations (chinoise ou israélienne) [Chang et al., 2004,Martinez-Borra et al., 2005,Chang et al., 2006]. De nombreuses études sur différentes populations (américaine, européenne, et chinoise), réaffirment, en plus, l'existence d'un fort déséquilibre de liaison entre le gène HCR et l'allèle HLA-Cw6 [Chia et al., 2001,O'Brien et al., 2001,Asumalahti et al., 2002,Chang et al., 2004,Helms et al., 2005,Chang et al., 2006,Suomela et al., 2007]. Une étude épidémiologique sur 379 patients finlandais montre que les allèles à risque HCR*WWCC (62%) et HLA-Cw6 (54%) sont associés aux mêmes caractéristiques cliniques. Par exemple, les patients porteurs de l'haplotype à risque HCR ont un âge de début plus précoce, ont généralement une infection de la gorge comme facteur déclenchant, des lésions de type psoriasis en gouttes et une histoire familiale de psoriasis plus importante que les patients non porteurs de cet haplotype. De plus, les patients non porteurs de cet haplotype ont des psoriasis plus graves de type pustulaire ou érythrodermique [Suomela et al., 2007].
). Les trois premiers variants semblent intervenir dans la structure secondaire de la protéine [Asumalahti et al., 2002]. Un ou l'ensemble de ces variants est montré(s) également associé(s) avec la maladie, et particulièrement avec le psoriasis de type I, dans d'autres populations (chinoise ou israélienne) [Chang et al., 2004,Martinez-Borra et al., 2005,Chang et al., 2006]. De nombreuses études sur différentes populations (américaine, européenne, et chinoise), réaffirment, en plus, l'existence d'un fort déséquilibre de liaison entre le gène HCR et l'allèle HLA-Cw6 [Chia et al., 2001,O'Brien et al., 2001,Asumalahti et al., 2002,Chang et al., 2004,Helms et al., 2005,Chang et al., 2006,Suomela et al., 2007]. Une étude épidémiologique sur 379 patients finlandais montre que les allèles à risque HCR*WWCC (62%) et HLA-Cw6 (54%) sont associés aux mêmes caractéristiques cliniques. Par exemple, les patients porteurs de l'haplotype à risque HCR ont un âge de début plus précoce, ont généralement une infection de la gorge comme facteur déclenchant, des lésions de type psoriasis en gouttes et une histoire familiale de psoriasis plus importante que les patients non porteurs de cet haplotype. De plus, les patients non porteurs de cet haplotype ont des psoriasis plus graves de type pustulaire ou érythrodermique [Suomela et al., 2007].
Des études supplémentaires sur l'expression génique de HCR indiquent une expression forte du gène seulement dans les kératinocytes basales (au niveau nucléaire et cytoplasmique) des lésions psoriasiques [Asumalahti et al., 2000]. Ils montrent aussi un profil d'expression différent selon le type de peau. Pour les peaux saines, une expression uniforme dans les kératinocytes basales est observée contrairement aux peaux lésées, où l'expression est plus faible dans les kératinocytes basales prolifératifs. Ceci suggère un rôle de HCR dans la régulation de la différenciation ou de la prolifération des cellules épidermiques [Asumalahti et al., 2002]. Pour étudier la fonction du gène HCR, des souris transgéniques sur-exprimant l'haplotype sauvage ou l'haplotype à risque HCR*WWCC dans les kératinocytes basales ont été générés. Bien qu'aucun phénotype particulier n'ait été observé chez ces souris transgéniques, des différences dans le profil d'expression génique, ressemblant à celles observées chez les patients, ont été révélées. Par exemple, chez les souris sur-exprimant l'haplotype à risque HCR, des gènes intervenant dans la prolifération ou la différenciation terminale des kératinocytes (comme les cytokératines 6, 16 et 17) et aussi dans la formation de l'enveloppe cellulaire cornifiée (tels que les SPRRs (small proline-rich proteins) et la transglutaminase TGM3) sont sur-exprimés [Elomaa et al., 2004].
L'ensemble de ces données tend à considérer ce gène comme un gène candidat du PSORS1, parfois indépendamment du gène HLA-C [Oka et al., 1999,Nair et al., 2000,Martinez-Borra et al., 2005,Orru et al., 2005] et du gène CDSN [Martinez-Borra et al., 2005]. Néanmoins, même en présence d'une forte association de la maladie avec le gène HCR, plusieurs études sur diverses populations (Europe, USA, Chine) semblent en désaccord avec le rôle possible de ce gène seul dans la maladie en raison du fort déséquilibre de liaison entre les gènes HLA-C et HCR dans ces populations, rendant difficile de différencier les activités réciproques de chacun des gènes [Asumalahti et al., 2000,Chia et al., 2001,O'Brien et al., 2001,Asumalahti et al., 2002,Veal et al., 2002,Helms et al., 2005,Nair et al., 2006,Fan et al., 2008].
Le deuxième gène le plus étudié au locus 6p21 (PSORS1) est la cornéodesmosine (CDSN, S gene). La cornéodesmosine est une protéine essentielle impliquée dans les structures intracellulaires de la couche cornée, les cornéodesmosomes. Cette protéine est synthétisée lors des dernières étapes de la différenciation des kératinocytes et semble avoir un rôle dans la cohésion entre les cellules épidermiques de la couche cornée ainsi que dans le processus de desquamation via les cornéodesmosomes. La protéolyse de certains composants cornéodesmosomaux comme la cornéodesmosine permettrait le détachement des cornéocytes superficiels [Zhou and Chaplin, 1993,Simon et al., 1997,Guerrin et al., 1998]. De nombreuses études montrent une association significative entre le gène CDSN polymorphe et le psoriasis dans la population caucasienne. Lors d'une étude TDT réalisée sur 152 trios anglais, trois SNPs non-synonymes (CDSN-619T, -1240G, -1243C dans l'exon 2, nommé CDSN*5) sont associés avec le psoriasis (P=3x10![]() ), apparemment indépendamment de l'allèle HLA-Cw6 [Allen et al., 1999]. D'autres études confirment l'association du gène CDSN avec le psoriasis dans la population européenne lors d'études TDT (CDSN*5_CDSN-1236T: 153 trios, P=8.5x10
), apparemment indépendamment de l'allèle HLA-Cw6 [Allen et al., 1999]. D'autres études confirment l'association du gène CDSN avec le psoriasis dans la population européenne lors d'études TDT (CDSN*5_CDSN-1236T: 153 trios, P=8.5x10![]() et CDSN-619T-1236T-1243C, nommé CDSN*TTC: 52 familles, P=0.003) ou lors d'études cas-témoins (CDSN*TTC:161 vs 184, P=8.3x10
et CDSN-619T-1236T-1243C, nommé CDSN*TTC: 52 familles, P=0.003) ou lors d'études cas-témoins (CDSN*TTC:161 vs 184, P=8.3x10![]() , CDSN-1243C: 235 vs 374) respectivement [Tazi Ahnini et al., 1999,Schmitt-Egenolf et al., 2001,Ameen et al., 2005,Orru et al., 2005]. Dans le dernier cas, l'allèle CDSN-1243*C est significativement associé avec la maladie, en particulier avec le psoriasis de type I, 65% des 235 patients sont porteurs de l'allèle C alors que seulement 47% des 374 témoins le sont (P=2x10
, CDSN-1243C: 235 vs 374) respectivement [Tazi Ahnini et al., 1999,Schmitt-Egenolf et al., 2001,Ameen et al., 2005,Orru et al., 2005]. Dans le dernier cas, l'allèle CDSN-1243*C est significativement associé avec la maladie, en particulier avec le psoriasis de type I, 65% des 235 patients sont porteurs de l'allèle C alors que seulement 47% des 374 témoins le sont (P=2x10![]() ). Un fort déséquilibre de liaison entre le gène CDSN et un autre allèle associé, le HLA-Cw6, est aussi observé (36% versus 10%, P=8x10
). Un fort déséquilibre de liaison entre le gène CDSN et un autre allèle associé, le HLA-Cw6, est aussi observé (36% versus 10%, P=8x10![]() ) [Tazi Ahnini et al., 1999]. Un constat similaire est obtenu avec des haplotypes longs contenant CDSN*5 dans d'autres études caucasiennes/européennes [Jenisch et al., 1999,Capon et al., 2003,Orru et al., 2005]. L'association de ce gène avec le psoriasis, particulièrement avec le psoriasis de type I, est aussi retrouvée dans la population chinoise [Chang et al., 2006]. Une étude sur 103 patients caucasiens atteints de psoriasis en gouttes suggère que ce gène intervient aussi dans la pathogenèse dans d'autres formes cliniques de psoriasis, et de manière indépendante de l'allèle HLA-Cw6 [Tazi-Ahnini et al., 1999].
) [Tazi Ahnini et al., 1999]. Un constat similaire est obtenu avec des haplotypes longs contenant CDSN*5 dans d'autres études caucasiennes/européennes [Jenisch et al., 1999,Capon et al., 2003,Orru et al., 2005]. L'association de ce gène avec le psoriasis, particulièrement avec le psoriasis de type I, est aussi retrouvée dans la population chinoise [Chang et al., 2006]. Une étude sur 103 patients caucasiens atteints de psoriasis en gouttes suggère que ce gène intervient aussi dans la pathogenèse dans d'autres formes cliniques de psoriasis, et de manière indépendante de l'allèle HLA-Cw6 [Tazi-Ahnini et al., 1999].
Sur le plan fonctionnel, une étude montre qu'un allèle de CDSN (971T), présent sur les haplotypes longs associés au psoriasis, entraîne une augmentation de la stabilité de l'ARNm [Capon et al., 2004a]. Puis, l'expression de CDSN semble être altérée de manière générale dans les lésions psoriasiques. Son expression est en effet élevée au niveau des couches épineuse et cornée au lieu d'être limitée à l'interface entre les couches granuleuse et cornée comme dans le cas d'une peau saine ou non lésée [Allen et al., 2001].
Néanmoins, d'autres études (Japon, Chine, Thaïlande, Europe) semblent en désaccord avec le rôle possible de ce gène dans la maladie, même avec l'existence d'un très fort DL avec HLA-Cw6 [Ishihara et al., 1996,Asumalahti et al., 2000,Enerback et al., 2000,Chang et al., 2003,Romphruk et al., 2003,Ameen et al., 2005,Helms et al., 2005,Martinez-Borra et al., 2005].
La localisation exacte du gène de PSORS1 reste controversée due à l'existence d'un fort déséquilibre de liaison dans la sous-région de classe I du système HLA, caractérisée par un taux de recombinaison de 2.3 fois inférieur au taux moyen sur le génome [Walsh et al., 2003]. Cela expliquerait les nombreuses associations observées entre le psoriasis et plusieurs gènes de la région, souvent de magnitude comparable à celle observée pour le gène initialement décrit HLA-C, puisqu'ils sont, comme précédemment exposé, eux-mêmes en déséquilibre de liaison entre eux.
Le gène HLA-C, et particulièrement l'antigène de classe I HLA-Cw6, reste néanmoins le meilleur candidat comme gène de prédisposition du PSORS1.
Depuis une vingtaine d'années, de nombreuses études sur différentes populations ont montré une forte association avec l'allèle HLA-Cw6 et le psoriasis [Tiilikainen et al., 1980,Elder et al., 1994,Mallon et al., 1999]. Environ 45% des personnes de type caucasien atteintes de psoriasis portent l'allèle HLA-Cw6 alors que cet allèle n'a qu'une fréquence de 10% dans la population générale [Trembath et al., 1997]. Cette association est particulièrement importante chez les patients avec un psoriasis à début précoce et un psoriasis en gouttes [Tiilikainen et al., 1980,Henseler and Christophers, 1985,Enerback et al., 1997,Mallon et al., 1997,Mallon et al., 1999,Gudjonsson et al., 2002,Szczerkowska-Dobosz et al., 2004]. Dans l'une des études, 85% des patients avec un psoriasis à début précoce (avant l'âge de 40 ans) ont au moins un allèle HLA-Cw6, comparé au 15% des patients atteints après 40 ans [Henseler and Christophers, 1985].
Les caractéristiques cliniques ou le risque de développer la maladie varient selon le type HLA du patient. Les patients porteurs du HLA-Cw602 ont des symptômes plus étendus et parfois plus sévères [Gudjonsson et al., 2002,Szczerkowska-Dobosz et al., 2004]. Ils sont également plus sensibles au phénomène de Koebner et ont une réponse plus favorable à la lumière du soleil [Gudjonsson et al., 2002]. Les patients portant HLA-Cw6 ont un risque augmenté de 10 à 20 fois de développer un psoriasis et les patients homozygotes pour cet allèle ont 2,5 fois plus de risque que les patients hétérozygotes [Gudjonsson et al., 2003]. En revanche, les défauts de l'ongle et l'arthrite psoriasique sont plus fréquents chez les patients non porteurs de l'allèle HLA-Cw6 [Gudjonsson et al., 2002].
Diverses études indiquent que PSORS1 est proche du gène HLA-C et distinct de la région contenant CDSN et HCR [Veal et al., 2002,Helms et al., 2005]. En effet, deux SNPs (nommés SNP7 et SNP9) présents à environ 5,7 et 10 kb en amont du codon d'initiation du gène HLA-C sont trouvés fortement associés avec le psoriasis, en comparaison avec d'autres SNPs de la région [Veal et al., 2002]. Des haplotypes contenant en majorité l'allèle HLA-Cw6 sont également préférentiellement transmis aux individus atteints dans une étude européenne. Commun à ces haplotypes, un autre variant semble de fonction intéressante puisqu'il entraîne la création d'un site de fixation pour le facteur de transcription RUNX et qu'il est en complet DL avec les deux polymorphismes en amont du gène HLA-C [Helms et al., 2005].
D'autres études de la région PSORS1 de 300kb, en combinant l'analyse d'haplotypes avec celle des séquences, ont récemment identifié HLA-Cw6 comme étant l'allèle causal [Enerback et al., 2000,Chang et al., 2003,Elder, 2006,Nair et al., 2006,Fan et al., 2008]. Une étude récente compare les séquences (environ 300kb) de 7 chromosomes afin d'identifier un variant ou une combinaison de variants qui serait présent uniquement sur les deux chromosomes à risque (HLA-B57-Cw6 et HLA-B50-Cw6) . Seulement deux gènes dans cet intervalle, HLA-C et CDSN, ont des allèles spécifiques aux chromosomes à risque qui affectent les protéines correspondantes. Or, une étude d'association sur la totalité des familles caucasiennes confirme que les allèles HLA-Cw6 et CDSN*TTC sont fortement associés avec le psoriasis. Une analyse dans les familles, de 76 haplotypes recombinants du chromosome à risque ancestral et porteurs soit de HLA-Cw6 ou soit de CDSN*TTC, montre que seuls les haplotypes HLA-Cw6+/CDSN*TTC- sont significativement associés avec la maladie et que par déduction, la région candidate est réduite à un segment de 158 kb excluant tous les gènes exceptés HLA-C et HCG27.
Ceci est confirmé par une analyse des familles chinoises atteintes de psoriasis. En effet, HLA-Cw6 est l'allèle du gène majeur produisant une molécule HLA de classe I, importante de par son implication dans la présentation de l'antigène aux cellules immunes (cellules T). Ainsi, l'antigène correspondant doit avoir un rôle important dans la maladie et sa connaissance pourrait être utilisée pour le développement d'un médicament.
Cependant, l'allèle HLA-Cw6 ne serait pas le seul allèle à risque dans le locus PSORS1 [Fan et al., 2008]. Cette hypothèse établie par de nombreuses études pourrait expliquer les différences observées lors de la cartographie fine de ce locus par plusieurs équipes [Schmitt-Egenolf et al., 2001,Capon et al., 2004a,Martinez-Borra et al., 2005] (Figure 1.14Cartographie de la région PSORS1 montrant la position des gènes connus ainsi que les régions candidates minimales pour contenir le gène causal identifiés lors des différentes études). Puisque le psoriasis est une "maladie complexe", il est possible qu'il y ait plus qu'un gène de prédisposition au psoriasis dans le locus PSORS1. Ces différences entre les études peuvent donc être dues à l'existence d'une hétérogénéité génétique entre les populations avec différents haplotypes à risque ou différents allèles à risque. L'allèle majeur HLA-Cw6 n'est présent que dans 40-80% des cas et sa prévalence dans la population générale n'est que de 10%, impliquant des facteurs ou des interactions de facteurs environnementaux et/ou génétiques supplémentaires et différentes selon les populations [Nair et al., 2000,Bowcock and Cookson, 2004]. Ainsi, il peut exister d'autres gènes capables de modifier le risque lié à HLA-Cw6 dans la région HLA. Par exemple, différents haplotypes qui portent l'allèle HLA-Cw6 avec différents allèles HLA-B (B57-Cw6, B13-Cw6 and B37-Cw6) confèrent un risque différent au psoriasis [Jenisch et al., 1998].
Le fait que PSORS1 ait une faible pénétrance (estimée à 15%) indique l'implication d'autres facteurs. Ces facteurs peuvent d'ailleurs conférer un risque de manière indépendante ou dépendante du locus PSORS1. Depuis quelques années, après la découverte d'une vingtaine de loci potentiels, les chercheurs tentent d'identifier les gènes, et les allèles correspondants prédisposant au psoriasis. Trois de ces régions sont d'ailleurs décrites par la suite et sont examinées en détail plus loin dans ce mémoire. Cependant, comme attendu pour une "maladie complexe", peu de gènes sont rapportés comme étant associés au psoriasis (Tableau 1.11Gènes et variants associés avec le psoriasis retrouvés dans au moins une étude).