



Next: Les gènes codant des
Up: Gènes «non associés» au
Previous: Gènes «non associés» au
Contents
Comme détaillé dans l'introduction, de nombreuses études réalisées sur différentes populations (USA, Suède, Irlande, Chine, Taiwan, Maroc) ont permis d'identifier la région 17q25 comme une région candidate forte pour le psoriasis (PSORS2) [Tomfohrde et al., 1994,Nair et al., 1997,Enlund et al., 1999a,Samuelsson et al., 1999,Zheng et al., 2003,Hwu et al., 2005,Birnbaum et al., 2006].
Des études de liaison (non paramétrique) suivies d'association (TDT) réalisées sur cette région de linkage (17q23-25) avec 242 familles nucléaires européennes, ont conduit à affiner la zone d'intérêt et à identifier deux régions significativement associées avec le psoriasis, l'une au niveau des gènes SLC9A3R1 et NAT9 et l'autre, plus distal (6 Mb), au niveau du gène RAPTOR [Helms et al., 2003,Speckman et al., 2003]. Dans la région SLC9A3R1/ NAT9, 5 variants non codants (rs745318, rs734232, rs895691, rs12797, rs2305214) sont fortement associés avec la maladie (P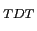 <0.005).
<0.005).
Les gènes SLC9A3R1/NAT9
SLC9A3R1 (solute carrier family sodium (Na+)/hydrogen (H+) exchanger 9, isoform 3 regulatory factor 1) code une phosphoprotéine contenant deux domaines PDZ (permettant les interactions protéine-protéine). Cette protéine est impliquée, entre autres dans la biologie de la membrane épithéliale en liant les protéines de membrane au cytosquelette, permettant ainsi la compartimentation correcte des protéines dans le domaine apical des cellules épithéliales. Elle aurait aussi un rôle de régulateur négatif de la formation des synapses immunes (interaction entre les cellules immunitaires) et de l'activation des cellules T en fixant le radeau lipidique (Cbp/PAG) au cytosquelette (ERM). Après reconnaissance de l'antigène par les cellules T, l'activation complète des cellules T nécessite une réorganisation du cytosquelette afin de recruter des molécules nécessaires à la réponse immune dans les radeaux lipidiques au niveau de la synapse. Cette protéine est exprimée dans la peau, plus fortement dans la couche granulaire mais aussi dans les cellules T. Elle est sous-régulée dans les cellules T actives. Ces observations sont indépendantes de l'état (sain ou malade) de la peau [Helms et al., 2003].
NAT9 code une protéine, qui est le membre 9 de la famille des N-acétyltransférases, de fonction inconnue. La protéine est exprimée dans les cellules T actives ou non, dans les kératinocytes quel que soit le type de peau (peau saine ou peau psoriasique et saine du patient) ou l'individu étudié (malade ou sain). La plus forte expression est observée dans de nombreuses cellules cancéreuses (ovaire, sein) [Helms et al., 2003].
Les deux gènes semblent être de bons candidats pour le psoriasis, d'autant plus qu'un des variants associés (rs734232), situé entre les deux gènes, est fonctionnellement intéressant. En effet, il abolit un site putatif de liaison du facteur de transcription RUNX1 (Runt-related transcription factor 1) in vivo et in vitro [Helms et al., 2003]. De plus, deux autres variants introniques altérant aussi un site de liaison pour RUNX1 au niveau des gènes PDCD1 et SLC22A4 ont également été montrés associés à deux autres maladies auto-immunes, lupus aigüe disséminé (SLE) et polyarthrite rhumatoïde (RA) [Prokunina et al., 2002,Tokuhiro et al., 2003]. RUNX1 est un facteur de transcription impliqué dans le développement des cellules hématopoïétiques. Il se pourrait que pour le psoriasis, la perte du site de liaison de RUNX1 perturbe la régulation de l'expression des gènes SLC9A3R1 et/ou NAT9 dans les cellules hématopoïétiques et par conséquent, le développement et l'activation des cellules T.
Néanmoins, la réplication indépendante de l'association dans diverses populations reste difficile. En effet, une étude d'association sur 233 trios anglais indépendants n'a pas permis de confirmer l'association des 5 SNPs (rs7420, rs734232, rs895691, rs12797, rs2305214) associés avec le psoriasis [Capon et al., 2004b]. De même, malgré leur preuve de liaison dans la région 17q25 à 1.7cM du coté distal du variant rs734232 (D17S785, NPL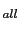 = 2.73, P=0.0002), aucune association entre 3 variants de SLC9A3R1 et/ou NAT9 et le psoriasis n'a été observée lors de l'étude de 517 familles allemandes et américaines [Stuart et al., 2005]. Une observation semblable a été décrite lors d'une étude du variant candidat (rs734232) réalisée sur des patients atteints de psoriasis vulgaire (PsV) allemands (300 trios et 281 cas) ainsi que sur 375 patients PsA [Huffmeier et al., 2005b]. Cependant, la stratification selon l'haplotype à risque de PSORS1 (HLA-Cw6) indique une association de l'allèle fréquent de ce SNP avec l'arthrite psoriasique chez les patients non-HLACw6 (P=0.0003) [Huffmeier et al., 2005b]. L'absence d'association avec ce SNP ou d'autres variants est aussi observée pour d'autres maladies auto-immunes comme l'arthrite psoriasique (PsA) ou la dermatite atopique (AD), ayant pourtant ce locus de prédisposition en commun. Cela a même été constaté dans différentes populations, par exemple, lors d'études cas-témoins sur des patients PsA provenant de l'île de Terre-Neuve (59 versus 238 témoins) [Butt et al., 2005] ou sur des patients AD japonais (114 versus 89) [Hosomi et al., 2005] ou encore lors d'étude TDT sur 426 familles européennes atteint d'AD [Morar et al., 2006]. De plus, lors d'une étude italienne (245 versus 150), aucune association entre deux SNPs (rs7420, rs734232) et PsA n'a été montrée, que le locus PSORS1 soit pris en compte ou non dans l'analyse [Giardina et al., 2006].
= 2.73, P=0.0002), aucune association entre 3 variants de SLC9A3R1 et/ou NAT9 et le psoriasis n'a été observée lors de l'étude de 517 familles allemandes et américaines [Stuart et al., 2005]. Une observation semblable a été décrite lors d'une étude du variant candidat (rs734232) réalisée sur des patients atteints de psoriasis vulgaire (PsV) allemands (300 trios et 281 cas) ainsi que sur 375 patients PsA [Huffmeier et al., 2005b]. Cependant, la stratification selon l'haplotype à risque de PSORS1 (HLA-Cw6) indique une association de l'allèle fréquent de ce SNP avec l'arthrite psoriasique chez les patients non-HLACw6 (P=0.0003) [Huffmeier et al., 2005b]. L'absence d'association avec ce SNP ou d'autres variants est aussi observée pour d'autres maladies auto-immunes comme l'arthrite psoriasique (PsA) ou la dermatite atopique (AD), ayant pourtant ce locus de prédisposition en commun. Cela a même été constaté dans différentes populations, par exemple, lors d'études cas-témoins sur des patients PsA provenant de l'île de Terre-Neuve (59 versus 238 témoins) [Butt et al., 2005] ou sur des patients AD japonais (114 versus 89) [Hosomi et al., 2005] ou encore lors d'étude TDT sur 426 familles européennes atteint d'AD [Morar et al., 2006]. De plus, lors d'une étude italienne (245 versus 150), aucune association entre deux SNPs (rs7420, rs734232) et PsA n'a été montrée, que le locus PSORS1 soit pris en compte ou non dans l'analyse [Giardina et al., 2006].
Malgré ces résultats contradictoires, nous voulions surtout essayer de répliquer, dans nos 45 familles, l'association observée lors de l'étude initiale faite par Helms et ses collaborateurs en 2003, entre le psoriasis et 5 variants non codants présents au niveau des gènes SLC9A3R1 et NAT9 (rs7420, rs734232, rs895691, rs12797, rs2305214), dont le variant abolissant le site de liaison à RUNX1. L'ensemble des résultats obtenus, lors de l'analyse par FBAT des 5 SNPs, ainsi que des haplotypes correspondants, sont exposés dans les tableaux 3.20 Identification et test d'association des TagSNPs de SLC9A3R1/NAT9 sur le Lot I par FBAT et 3.21Analyse d'association des haplotypes de SLC9A3R1/NAT9 sur le Lot I par FBAT. Aucune association n'a été observée entre le psoriasis et les 5 variants ainsi que lors de leur analyse après la stratification des familles selon le facteur de risque de PSORS1 (Tableau 3.22Analyse d'association des TagSNPs de SLC9A3R1/NAT9 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I). Cette absence d'association du psoriasis avec chacun des cinq variants dans nos 45 familles françaises est en rapport avec d'autres études réalisées sur différentes populations de tailles diverses (Angleterre, USA/Allemagne) [Capon et al., 2004b,Stuart et al., 2005,Huffmeier et al., 2005b].
L'absence de réplication sur différents cohortes indépendantes impose une certaine prudence quant à l'implication de ces variants, en particulier du SNP intervenant dans la fixation du facteur de transcription RUNX1, dans la pathogenèse du psoriasis. Dans notre population, ces variants ne semblent pas intervenir comme
Table 3.20:
Identification et test d'association des TagSNPs de SLC9A3R1/NAT9 sur le Lot I par FBAT
|
|
|
|
|
|
|
|
FBAT Lot I |
|
|
|
|
|
|
|
rs7420 |
3'NG |
|
6691487 |
A>G |
45.8 |
A |
51 |
100 |
-0.195 |
0.85 |
|
|
rs734232 |
3'NG |
|
6692003 |
G>A |
44.9 |
A |
41.7 |
99 |
-0.266 |
|
|
rs895691 |
3'NG |
|
6692572 |
G>T |
44 |
A |
41.7 |
102 |
-0.159 |
|
|
rs12797 |
3' UTR |
|
6693265 |
C>T |
45.8 |
A |
42 |
99 |
-0.395 |
|
|
rs2305214 |
Intron 4 |
|
6695242 |
C>T |
49.1 |
A |
41 |
100 |
-0.372 |
 associé avec le psoriasis (Helms et al., 2003), associé avec le psoriasis (Helms et al., 2003),
 NM_015654, NM_015654,  NT_010641.15 NT_010641.15 |
|
|
Table 3.21:
Analyse d'association des haplotypes de SLC9A3R1/NAT9 sur le Lot I par FBAT
| |
Lot I |
| |
|
MAF |
P |
| H1 |
AATTT |
45.5 |
0.36 |
| H2 |
GGGCC |
45.5 |
0.62 |
| H3 |
AGGCC |
7.7 |
0.27 |
Table 3.22:
Analyse d'association des TagSNPs de SLC9A3R1/NAT9 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
| |
|
Lot I |
| |
|
|
Stratifié selon: |
| |
|
|
|
présence HLA-Cw6 |
absence HLA-Cw6 |
| |
|
MAF |
P |
N |
Z |
P |
N |
| rs7420 |
A>G |
51 |
0.85 |
59 |
0.253 |
0.80 |
53 |
-0.596 |
0.55 |
| rs734232 |
G>A |
41.7 |
0.79 |
56 |
-0.481 |
0.63 |
55 |
0.149 |
0.88 |
| rs895691 |
G>T |
41.7 |
0.87 |
58 |
-0.523 |
0.60 |
57 |
0.352 |
0.73 |
| rs12797 |
C>T |
42 |
0.69 |
56 |
-0.682 |
0.50 |
55 |
0.183 |
0.86 |
| rs2305214 |
C>T |
41 |
0.71 |
57 |
-0.712 |
0.48 |
55 |
0.253 |
0.80 |
|
facteurs de prédisposition au psoriasis. Même si aucune liaison à ce locus n'a été identifiée dans nos familles, nous ne pouvons pas exclure entièrement le gène en raison de l'absence d'analyse globale du gène.
Le gène RAPTOR
Comme décrit précédemment, un second locus de prédisposition au psoriasis a été identifié sur le chromosome 17q25, proche du marqueur initialement lié D17S784 et du gène RAPTOR (Regulatory associated protein of mTOR). Ce gène code pour une protéine régulatrice qui s'associe à la protéine sérine thréonine kinase mTOR (mammalian target of rapamycine). En réponse à des signaux prolifératifs tels que les cytokines ou des facteurs de croissance (Insuline, IGF1..), la protéine mTOR joue un rôle clé dans la régulation traductionnelle de protéines impliquées dans de multiples voies de signalisation dont le contrôle du cycle cellulaire (VEGF,...) ou le transfert du signal d'activation et de prolifération de certaines cellules dont les lymphocytes T et les lymphocytes B. De par sa fonction et sa localisation, la protéine RAPTOR semble être un bon candidat pour le psoriasis.
De plus, des SNPs introniques du gène RAPTOR ont été montrés associés avec le psoriasis dans des familles américaines et anglaises [Helms et al., 2003,Capon et al., 2004b]. En effet, sur 233 trios anglais indépendants, une étude d'association de 3 SNPs de l'intron 3 de ce gène, initialement montrés associés dans l'étude de Helms (2003) (rs1564864, rs2019154, rs869190), a confirmé l'association de ce gène avec le psoriasis, plus particulièrement avec le SNP rs2019154 (P=0.027). Une stratification des trios selon leur provenance familiale (116 trios restants) a révélé, de manière plus significative, l'association de trois variants introniques avec la maladie (rs1564864 (P=0.031), rs2019154 (P=0.008), rs869190 (P=0.05) [Capon et al., 2004b]. Cependant, cette association n'a pas été confirmée lors de l'analyse de ces variants dans 517 familles allemandes et américaines atteintes de psoriasis [Stuart et al., 2005]. Des études sur l'implication de ces marqueurs dans d'autres maladies proches du psoriasis sur le plan clinique, comme le PsA et l'AD, ne montrent également aucune association probante. Le manque d'association de deux SNPs (rs869190, rs1561946) a aussi été remarqué lors d'une étude italienne cas-témoins sur le PsA (245 versus 150), même lorsque le locus PSORS1 est considéré [Giardina et al., 2006]. Pour l'AD, une autre maladie de la peau où le chromosome 17q est aussi une région candidate, ce gène ne semble pas intervenir dans la prédisposition à cette maladie [Morar et al., 2006].
A la vue de ces résultats contradictoires, nous avons entrepris de répliquer, dans nos 45 familles, l'association, observée lors de l'étude initiale faite par Helms et ses collaborateurs (2003), entre le psoriasis et 3 variants introniques présents dans le gène RAPTOR (rs1564864, rs2019154, rs869190). L'ensemble des résultats obtenus, lors de l'analyse par FBAT des 2 SNPs finalement génotypés ainsi que
Table 3.24:
Analyse d'association des haplotypes de RAPTOR sur le Lot I par FBAT
| |
Lot I |
| |
|
MAF |
P |
| H1 |
TG |
74.5 |
0.49 |
| H2 |
CT |
24.9 |
0.45 |
Table 3.25:
Analyse d'association des TagSNPs de RAPTOR dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
| |
|
Lot I |
| |
|
|
Stratifié selon: |
| |
|
|
|
présence HLA-Cw6 |
absence HLA-Cw6 |
| |
|
MAF |
P |
N |
Z |
P |
N |
| rs1564864 |
T>C |
23.4 |
0.48 |
54 |
0.138 |
0.89 |
49 |
0.900 |
0.37 |
| rs869190 |
G>T |
24.2 |
0.41 |
52 |
0.285 |
0.78 |
49 |
0.900 |
0.37 |
des haplotypes correspondants, sont exposés dans les tableaux 3.23Identification et test d'association des TagSNPs de RAPTOR sur le Lot I par FBAT et 3.24Analyse d'association des haplotypes de RAPTOR sur le Lot I par FBAT. Même lorsque PSORS1 est pris en compte, aucune association n'est observée entre le psoriasis et ces deux marqueurs (Tableau 3.25Analyse d'association des TagSNPs de RAPTOR dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I). Nos données contredisent les deux études anglaises et américaines, rapportant une association entre le psoriasis et RAPTOR [Helms et al., 2003,Capon et al., 2004b]. Néanmoins, ils sont en accord avec une étude plus récente réalisée sur un grand nombre de grandes familles allemandes et américaines [Stuart et al., 2005]. Comme expliqué précédemment, il serait intéressant de réaliser une étude globale du gène afin de l'exclure.
Nous pouvons cependant conclure que dans notre population, les deux variants de RAPTOR ne sont pas impliqués dans la liaison du locus PSORS2. D'ailleurs, plusieurs études plus récentes réalisées sur quelques familles identifient un autre gène potentiel pour PSORS2 de fonction inconnue, le gène ZNF750 situé du coté distal du gène RAPTOR (environ 2Mb), détaillé dans le chapitre Introduction [Hwu et al., 2005,Birnbaum et al., 2006,Yang et al., 2008]. Des études supplémentaires dans nos familles, par exemple l'étude complète du gène RAPTOR ou l'étude du nouveau gène d'intérêt ZNF750 permettraient de confirmer ou d'exclure ces gènes.




Next: Les gènes codant des
Up: Gènes «non associés» au
Previous: Gènes «non associés» au
Contents
anouar
2009-08-22