



Next: Le gène CYLD (CYLinDromatosis
Up: Gènes «associés» au psoriasis
Previous: Exemple particulier du gène
Contents
Un locus de prédisposition commun au psoriasis et à l'arthrite psoriasique a été identifié au niveau du bras long du chromosome 16 (16q) par différentes études de liaison (PSORS8) [Nair et al., 1997,Allen et al., 2003,Karason et al., 2003,Sagoo et al., 2004]. Cette région contient le gène CARD15, conférant une prédisposition à une maladie inflammatoire de la voie gastro-intestinale, la maladie de Crohn (CD), particulièrement avec trois variants (leu1007fsinsC (rs2066847, rs5743293), R702W (rs2066844) et G908R (rs2066845)) [Hugot et al., 2001,Ogura et al., 2001]. L'existence de ce locus commun entre ces trois maladies inflammatoires et la co-occurrence de ces maladies chez les malades [Lee et al., 1990] semblent suggérer que CARD15 serait un gène de prédisposition commun à ces trois maladies. D'ailleurs une étude sur des patients atteints d'arthrite psoriasique (PsA), venant de l'île de Terre Neuve, montre une association avec un des trois variants de CARD15 connus pour leur prédisposition à la maladie de Crohn (CD) [Rahman et al., 2003]. Cependant, aucune association avec ces trois variants n'a été confirmée lors des nombreuses études réalisées sur différentes populations, plus ou moins grandes, atteintes d'arthrite psoriasique (italienne, anglaise, allemande, américaine) [Giardina et al., 2004,Ho et al., 2005,Lascorz et al., 2005,Jenisch et al., 2006]. Ce manque d'association avec au moins l'un de ces trois variants est aussi observé dans le cas de l'étude du psoriasis, réalisée sur des populations caucasiennes (italiennes, anglaises et allemandes) [Nair et al., 2001,Borgiani et al., 2002,Young et al., 2003,Plant et al., 2004,Lascorz et al., 2005]. Une seule étude, certes négative, a fait une analyse globale du gène CARD15 dans la prédisposition au psoriasis, c'est-à-dire l'étude des TagSNPs et non exclusivement, l'étude d'un ou plusieurs des trois variants connus [Plant et al., 2004].
Etant donné ces résultats contradictoires, il était intéressant d'étudier ce gène, surtout de par sa fonction. Le gène CARD15 code une protéine intra-cytoplasmique relativement ubiquitaire, exprimée dans les monocytes/macrophages mais également dans les cellules épithéliales, les cellules dendritiques et les lymphocytes T. Cette protéine contient deux domaines de recrutement et d'activation des caspases situés en N-terminal (CARD, caspase-recruitment domain), un domaine de liaison aux nucléotides (NBD, nucleotide-binding domain) dans la partie médiane et un domaine C-terminal composé de motifs répétés, riches en leucines (LRRs, leucin-rich repeats). Les principales mutations impliquées dans la maladie de Crohn sont situées dans le dernier domaine, LRR (Figure 3.4Représentation de la structure du gène et de la protéine CARD15 et localisation des principales mutations associées à la maladie de Crohn).
Le rôle fonctionnel des LRRs n'est actuellement pas encore totalement élucidé. Ils interviendraient dans la reconnaissance de composants bactériens comme le muramyl dipeptide présent dans le peptidoglycan (PGN), constituant essentiel de la paroi des bactéries. La reconnaissance des composants bactériens déclenche une cascade d'événements dans le cytoplasme de la cellule aboutissant à l'activation de la voie de transduction du signal NF-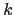 B (Figure 3.5Fonction proposée de CARD15 dans la régulation de l'inflammation par l'intermédiaire de l'activation de la voie NF-$k$B en réponse à différents composants bactériens) avec la migration de NF-
B (Figure 3.5Fonction proposée de CARD15 dans la régulation de l'inflammation par l'intermédiaire de l'activation de la voie NF-$k$B en réponse à différents composants bactériens) avec la migration de NF-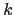 B dans le noyau cellulaire et la transcription de nombreux gènes dont ceux codant des cytokines inflammatoires (de type 1), de chimiokines, responsables de l'inflammation. La protéine CARD15 pourrait donc intervenir dans la régulation de l'inflammation induite par les bactéries, c'est-à-dire, dans la protection de l'hôte contre les pathogènes. D'ailleurs, des études ont mis en évidence que la présence des 3 principales mutations de CARD15 observées dans la maladie de Crohn provoquait in vitro un défaut d'activation de la voie NF-
B dans le noyau cellulaire et la transcription de nombreux gènes dont ceux codant des cytokines inflammatoires (de type 1), de chimiokines, responsables de l'inflammation. La protéine CARD15 pourrait donc intervenir dans la régulation de l'inflammation induite par les bactéries, c'est-à-dire, dans la protection de l'hôte contre les pathogènes. D'ailleurs, des études ont mis en évidence que la présence des 3 principales mutations de CARD15 observées dans la maladie de Crohn provoquait in vitro un défaut d'activation de la voie NF-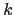 B, partiel ou total, surtout en réponse à la stimulation par des agents bactériens (LPS ou PGN) [Ogura et al., 2001,Chamaillard et al., 2003]. De plus, une étude in vitro sur l'homologue murin de CARD15 (80% d'homologie en acide aminé), le gène Nod2, montre que deux
B, partiel ou total, surtout en réponse à la stimulation par des agents bactériens (LPS ou PGN) [Ogura et al., 2001,Chamaillard et al., 2003]. De plus, une étude in vitro sur l'homologue murin de CARD15 (80% d'homologie en acide aminé), le gène Nod2, montre que deux
Figure 3.4:
Représentation de la structure du gène et de la protéine CARD15 et localisation des principales mutations associées à la maladie de Crohn (D'après [Lamoril et al., 2007])
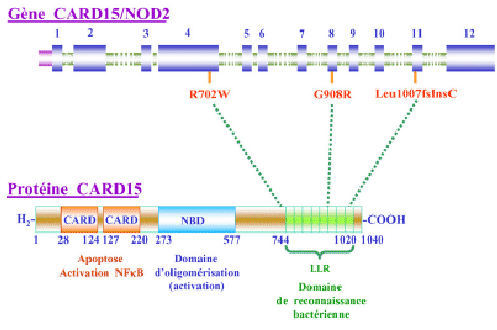 |
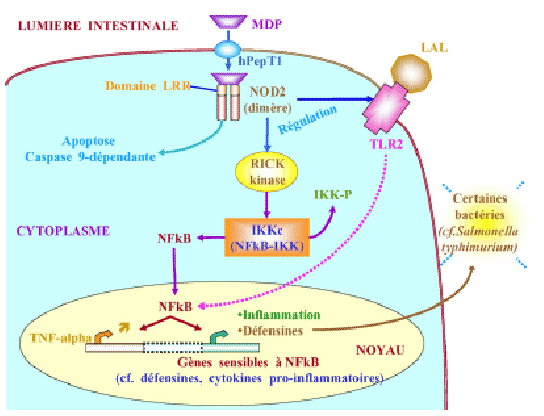
Figure 3.5:
Fonction proposée de CARD15 dans la régulation de l'inflammation par l'intermédiaire de l'activation de la voie NF-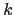 B en réponse à différents composants bactériens
B en réponse à différents composants bactériens
Après pénétration dans le cytoplasme du muramyldipeptide (MDP), constituant du peptidoglycan des parois bactériennes, à l'aide d'un transporteur, le MDP interagit avec le motif LRR de la protéine CARD15. Cette reconnaissance du motif MDP par CARD15 provoquerait, avec l'intervention de différents intermédiaires, l'activation de la voie NF-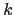 B, aboutissant à la sécrétion de peptides antimicrobiens, tels que les défensines et les cytokines pro-inflammatoires. (D'après [Lamoril et al., 2007])
B, aboutissant à la sécrétion de peptides antimicrobiens, tels que les défensines et les cytokines pro-inflammatoires. (D'après [Lamoril et al., 2007])
 |
mutations de Nod2 équivalentes aux mutations G908R et 3020insC responsables de la maladie de Crohn chez l'homme, mutations G908R et 3020insC responsables de la maladie de Crohn (CD) chez l'homme, provoquent une faible activation de NF-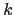 B en présence ou non de composants bactériens (LPS et PGN) [Ogura et al., 2003]. Ces observations sont cependant en contradiction avec des études in vivo et in vitro sur des modèles murins qui démontrent que la protéine CARD15 mutée ou déficiente permettrait l'augmentation de l'activation de NF-
B en présence ou non de composants bactériens (LPS et PGN) [Ogura et al., 2003]. Ces observations sont cependant en contradiction avec des études in vivo et in vitro sur des modèles murins qui démontrent que la protéine CARD15 mutée ou déficiente permettrait l'augmentation de l'activation de NF-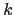 B et des réponses Th1 après fixation d'un peptidoglycan sur le récepteur TLR2 (Toll-like receptor 2) [Watanabe et al., 2004,Maeda et al., 2005]. Ce paradoxe n'est toujours pas résolu à ce jour mais de manière générale, CARD15 semble être un important régulateur de l'activation de NF-
B et des réponses Th1 après fixation d'un peptidoglycan sur le récepteur TLR2 (Toll-like receptor 2) [Watanabe et al., 2004,Maeda et al., 2005]. Ce paradoxe n'est toujours pas résolu à ce jour mais de manière générale, CARD15 semble être un important régulateur de l'activation de NF-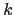 B et les mutations de CARD15 paraissent modifier les signaux de la réponse immunitaire innée (non spécifique) en réponse aux motifs bactériens, ce qui aboutirait à une incapacité du système immunitaire à contrôler l'infection bactérienne par une élimination moins efficace des bactéries qui conduirait à une inflammation chronique.
B et les mutations de CARD15 paraissent modifier les signaux de la réponse immunitaire innée (non spécifique) en réponse aux motifs bactériens, ce qui aboutirait à une incapacité du système immunitaire à contrôler l'infection bactérienne par une élimination moins efficace des bactéries qui conduirait à une inflammation chronique.
Puisque le psoriasis peut être déclenché par, entre autres, des infections bactériennes (psoriasis en gouttes) et que la majorité des études sur le psoriasis ne prenait en compte que les trois variants associés à CD et causant une altération dans la séquence peptidique, alors qu'il existe aussi d'autres polymorphismes associés, même introniques [Sugimura et al., 2003], nous avons entrepris de tester le gène entier par une approche "TagSNP". Pour cela, dans nos 45 familles, nous avons analysé, par FBAT, 6 TagSNPs choisis en fonction de la structure DL du gène obtenu par les données HapMap/dbSNP (Figure 3.6 Structure DL du gène CARD15) en les ajoutant à ceux associés aux maladies auto-immunes (rs2066844, rs2066845 et rs5743293 pour CD, rs2076752, rs1077861, rs3135500 pour l'asthme et la rhino-conjonctivite allergique) [Weidinger et al., 2005]. Les résultats obtenus dans notre lot I sont présentés dans le tableau 3.10 Identification et test d'association des TagSNPs de CARD15 sur le Lot I par FBAT.
Des associations sont identifiées entre le psoriasis et trois polymorphismes, rs2076752 dans la partie 5' non traduite, rs17221417 dans l'intron 2 et rs2066844 dans l'exon 4 avec un P de 0.005, de 0.01 et de 0.04 respectivement. Ormis rs2066844, les deux autres SNPs associés à CD n'ont pu être analysés dans nos familles. En effet, peu de nos familles nucléaires sont informatives pour permettre le test TDT du SNP rs2066845, qui a une fréquence de l'allèle rare de 1% seulement dans notre population. De plus, la délétion rs5743293 n'a pas pu être génotypé pour des raisons techniques. Cependant, une étude supplémentaire, en séquençant l'exon 11 sur 140 individus atteints indépendants et 94 témoins, ne montre aucune différence de la fréquence de l'allèle mineur de ce SNP dans les deux groupes. Par la suite, nous avons testé si des haplotypes du gène CARD15 pouvaient jouer un rôle dans la prédisposition au psoriasis. Etant donné l'existence d'un faible déséquilibre de liaison dans le gène, nous n'avons pu testé que la transmission préférentielle aux
Figure 3.6:
Structure DL du gène CARD15
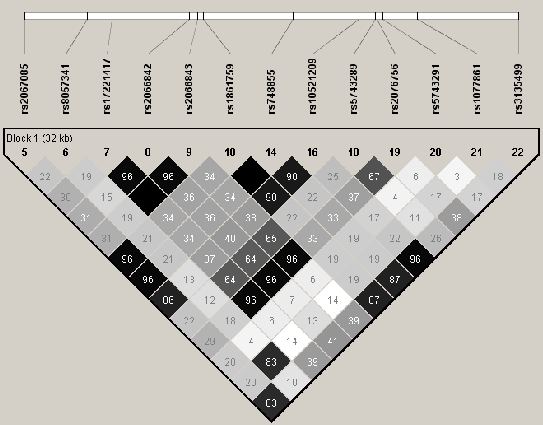 |
Table 3.10:
Identification et test d'association des TagSNPs de CARD15 sur le Lot I par FBAT
|
|
|
|
|
|
|
|
|
FBAT Lot I Lot I |
|
|
|
|
|
|
|
1 |
rs2076752 |
5' UTR |
|
4345295^96 |
G>A |
22 |
A |
33 |
92 |
-2.790 |
0.005 |
|
|
2 |
rs2111235 |
Intron 2 |
|
4348168 |
C>T |
25 |
D |
29 |
92 |
0.848 |
|
|
3 |
rs8057341 |
Intron 2 |
|
4352179 |
G>A |
25.8 |
H |
29.1 |
92 |
0.677 |
|
|
4 |
rs17221417 |
Intron 2 |
|
4353781 |
C>G |
35.8 |
H |
35.5 |
91 |
-2.447 |
|
|
5 |
rs2066842 |
Exon 4 |
P268S |
4358823 |
C>T |
36.2 |
H |
|
|
|
|
|
6 |
rs2066844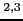 |
Exon 4 |
R702W |
4360125 |
C>T |
11 |
A |
7.4 |
31 |
-2.065 |
|
|
7 |
rs7203691 |
Intron 6 |
|
4367623 |
G>A |
36.4 |
D |
36.2 |
107 |
1.612 |
|
|
8 |
rs10521209 |
Intron 7 |
|
4369908 |
T>G |
40.8 |
H |
36.5 |
108 |
1.46 |
|
|
9 |
rs2066845 |
Exon 8 |
G908R |
4370739 |
G>C |
1.7 |
A |
1 |
6 |
*** |
|
|
10 |
rs5743291 |
Exon 9 |
V955I |
4371475 |
G>A |
10.8 |
H |
10.9 |
48 |
1.550 |
|
|
11 |
rs1077861 |
Intron 10 |
|
4373746 |
A>T |
25 |
A |
33.3 |
99 |
0.122 |
|
|
12 |
rs2066847 |
Exon 11 |
P1006fs |
4377977^78 |
->C |
0 |
A |
|
|
|
|
|
13 |
rs5743293 |
Exon 11 |
L1007fs |
4377980^81 |
->C |
2.5 |
A |
|
|
|
|
|
14 |
rs3135500 |
3' UTR |
|
4381085 |
G>A |
39.7 |
A |
37.3 |
108 |
1.063 |
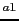 associé avec l'asthme ou la rhinoconjonctivite allergique (Weidinger et al., 2005), associé avec l'asthme ou la rhinoconjonctivite allergique (Weidinger et al., 2005),  associé avec la maladie de Crohn (Hugot et al., 2001; Ogura et al., 2001), associé avec la maladie de Crohn (Hugot et al., 2001; Ogura et al., 2001), 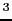 associé avec l'arthrite psoriasique (Rahman et al., 2003), associé avec l'arthrite psoriasique (Rahman et al., 2003),  NM_022162, NM_022162, 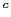 NT_010498.15, NT_010498.15, 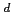 "***": aucun test réalisé en raison d'un faible nombre de familles informatives (N<10) "***": aucun test réalisé en raison d'un faible nombre de familles informatives (N<10)
|
|
|
individus atteints de toutes les combinaisons de paires de SNPs. Nombre de ces combinaisons sont trouvées associées au psoriasis (Table 3.11Analyse d'association des haplotypes de CARD15 sur le Lot I par FBAT). Nous pouvons observer une sous-transmission préférentielle des parents hétérozygotes aux enfants atteints de nombreux haplotypes (Z<0), avec une valeur de P variant de 0.001 à 0.04 après 1000000 permutations. Ces haplotypes protecteurs sont constitués d'au moins un des SNPs associés comme rs2076752, rs17221417 et rs2066844 ou, ayant une tendance à être associés à la maladie comme rs7203691, rs10521209, rs5743291, rs3135500. Les quelques haplotypes à risque (Z>0) associés au psoriasis sont dépendants de trois allèles à risque. L'allèle fréquent de rs2076752 (G) est présent sur des haplotypes de fréquences élevées (70%) ou plus basses (38%/10%) qui donnent, lors du test d'association, un P variant de 0.004 à 0.05. L'allèle fréquent de rs17221417 (C) est présent sur deux haplotypes de haute fréquence (70%) qui sont associés au psoriasis avec un P de 0.01. De plus, l'allèle rare de rs5743291 (A) cible deux haplotypes de 10% qui sont associés à la maladie avec un P proche de 0.03. Puisque la fréquence des haplotypes associés à la maladie est similaire à celle des allèles qui les représentent, cela semble montrer que ce sont les allèles fréquents, G et C, pour les deux premiers SNPs ainsi que l'allèle rare A de rs5743291 qui confèrent le risque pour le psoriasis. A part pour rs5743291, cette analyse haplotypique semble refléter les résultats obtenus lors de l'étude individuelle des SNPs dans le lot I. L'association des trois SNPs, identifiés précédemment avec la maladie, a pu être confirmée lors de l'étude avec la méthode "LNMs". En effet, nous observons une valeur de P de 0.003, 0.003 et 0.01 pour les polymorphismes rs2076752, rs17221417 et rs2066844 respectivement (Table 3.12 Analyse d'association des TagSNPs de CARD15 dans le Lot I avec la méthode "LNMs").
L'étude de deux marqueurs, rs2076752 et rs17221417, dans le lot II moins informatif, ainsi que celle dans les lots I et II combinés, suggère, même faiblement, l'association des deux polymorphismes avec le psoriasis (P=0.04 et P=0.06 respectivement) (Table 3.13Analyse d'association des TagSNPs de CARD15, suggestifs dans le Lot I, faite sur le Lot II et ainsi que sur l'ensemble des lots (Lot I et Lot II)). En revanche, pour rs2066844, nous observons une association avec la maladie dans la deuxième cohorte, équivalente à celle obtenue dans le premier lot. Cette association dans le premier lot est due à l'effet protecteur de l'allèle rare T alors que dans le deuxième lot, l'association est due à l'effet à risque de l'allèle T, ce qui entraîne l'absence d'association lors de l'étude des deux lots combinés.
Lors de la dernière étude, l'association de plusieurs variants (rs2076752, rs17221417, rs7203691, rs10521209, rs5743291 et rs3135500) avec le psoriasis est observée seulement lorsque nous analysons les personnes porteuses de l'allèle HLA-Cw6, indiquant ainsi une interaction de ces deux gènes dans la maladie (Table 3.14Analyse d'association des TagSNPs de CARD15 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I).
A la suite de ces résultats, aucune association probante n'a été détectée entre le psoriasis et les trois variants de CARD15 prédisposant à la maladie de Crohn (rs2066844, rs2066845 et rs5743293) dans nos familles françaises. Ceci semble confirmer les résultats précédemment obtenus sur le psoriasis réalisées sur d'autres
-1cm
Table 3.11:
Analyse d'association des haplotypes de CARD15 sur le Lot I par FBAT
| |
|
Lot I |
| |
|
MAF |
Z |
P |
| 1/2 |
AC |
29.2 |
-3.164 |
0.0016 |
0.0018 |
(0.0069) |
| 1/3 |
AG |
29.2 |
-3.198 |
0.0014 |
0.0012 |
(0.0060) |
| |
GC |
70.2 |
2.673 |
0.0075 |
0.0076 |
2(0.014) |
| |
AG |
28.3 |
-2.727 |
0.0064 |
0.0059 |
| 1/6 |
GC |
71.2 |
2.811 |
0.0050 |
0.0057 |
(0.0075) |
| 1/7 |
AG |
29.2 |
-2.725 |
0.0064 |
0.0070 |
(0.025) |
| 1/8 |
AT |
28.9 |
-2.789 |
0.0053 |
0.0042 |
(0.018) |
| 1/9 |
GG |
71.0 |
2.802 |
0.0051 |
0.0042 |
(0.0041) |
| |
AG |
29.1 |
-3.004 |
0.0027 |
0.0026 |
2(0.0045) |
| |
GA |
10.7 |
2.550 |
0.011 |
0.0093 |
| |
GA |
37.8 |
1.961 |
0.049 |
0.049 |
2(0.0068) |
| |
AA |
25.8 |
-2.535 |
0.011 |
0.0099 |
| 1/14 |
AG |
28.7 |
-2.738 |
0.0061 |
0.0061 |
(0.035) |
| 2/4 |
CG |
29 |
-2.728 |
0.0064 |
0.0064 |
(0.032) |
| 2/6 |
CT |
6 |
-2.551 |
0.011 |
0.015 |
(0.084) |
| 2/7 |
CG |
31.2 |
-2.783 |
0.0054 |
0.0053 |
(0.032) |
| 2/8 |
CT |
31.1 |
-2.842 |
0.0045 |
0.0038 |
(0.016) |
| |
CG |
56.4 |
-2.303 |
0.021 |
0.017 |
2(0.031) |
| |
CA |
10.4 |
2.092 |
0.036 |
0.035 |
| 2/14 |
CG |
32.3 |
-2.173 |
0.030 |
0.024 |
(0.14) |
| 3/4 |
GG |
28.9 |
-2.714 |
0.0066 |
0.0062 |
(0.033) |
| 3/6 |
GT |
6.1 |
-2.554 |
0.011 |
0.013 |
(0.079) |
| 3/7 |
GG |
30.5 |
-2.547 |
0.011 |
0.012 |
(0.021) |
| 3/8 |
GT |
30.4 |
-2.607 |
0.0091 |
0.0095 |
(0.038) |
| |
GG |
56.3 |
-2.296 |
0.022 |
0.020 |
2(0.031) |
| |
GA |
10.3 |
2.069 |
0.039 |
0.035 |
| 3/14 |
GG |
32.1 |
-2.041 |
0.041 |
0.040 |
(0.19) |
| |
CC |
70.7 |
2.552 |
0.011 |
0.012 |
2(0.013) |
| |
GT |
6.1 |
-2.125 |
0.034 |
0.026 |
| 4/7 |
GG |
29.6 |
-2.361 |
0.018 |
0.015 |
(0.060) |
| 4/8 |
GT |
29.6 |
-2.361 |
0.018 |
0.018 |
(0.060) |
| 4/9 |
CG |
70.5 |
2.457 |
0.014 |
0.013 |
(0.011) |
| |
GG |
29.7 |
-2.535 |
0.011 |
0.015 |
2(0.020) |
| |
CA |
10.3 |
2.233 |
0.026 |
0.022 |
| 4/11 |
GA |
26.1 |
-2.047 |
0.04 |
0.044 |
(0.028) |
| 4/14 |
GG |
29.4 |
-2.427 |
0.015 |
0.015 |
(0.051) |
| 6/7 |
TG |
6 |
-2.203 |
0.028 |
0.023 |
(0.070) |
| 6/8 |
TT |
6 |
-2.203 |
0.028 |
0.027 |
(0.084) |
| |
CG |
92.8 |
2.522 |
0.012 |
0.012 |
20.016) |
| |
TG |
5.9 |
-2.065 |
0.039 |
0.038 |
| 6/10 |
TG |
6 |
-2.092 |
0.036 |
0.044 |
(0.11) |
| 6/11 |
TA |
6 |
-2.542 |
0.011 |
0.013 |
(0.057) |
| 6/14 |
TG |
5.9 |
-2.134 |
0.033 |
0.030 |
(0.15) |
| 7/11 |
GA |
27.7 |
-2.236 |
0.025 |
0.030 |
(0.032) |
| 8/11 |
TA |
27.5 |
-2.003 |
0.045 |
0.050 |
(0.10) |
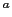 La valeur exacte de P du Z est calculée via la méthode de Monte-Carlo pour 1.000.000 de permutations pour chaque haplotype séparément (nommé "1 000 000 permutations P") et pour le test global (nommé "1 000 000 permutations P La valeur exacte de P du Z est calculée via la méthode de Monte-Carlo pour 1.000.000 de permutations pour chaque haplotype séparément (nommé "1 000 000 permutations P") et pour le test global (nommé "1 000 000 permutations P "). ").
|
|
Table 3.13:
Analyse d'association des TagSNPs de CARD15, suggestifs dans le Lot I, faite sur le Lot II et ainsi que sur l'ensemble des lots (Lot I et Lot II)
| |
Lot I |
Lot II |
Lot I + Lot II |
| |
MAF |
N |
Z |
P |
MAF |
N |
Z |
P |
MAF |
N |
Z |
| rs2076752 |
33 |
92 |
-2.79 |
0.005 |
31.3 |
65 |
0.33 |
0.74 |
32 |
156 |
-2.022 |
0.04 |
| rs17221417 |
35.5 |
91 |
-2.45 |
0.01 |
29.3 |
59 |
0.16 |
0.87 |
32.2 |
149 |
-1.917 |
0.06 |
| rs2066844 |
7.4 |
31 |
-2.07 |
0.04 |
7.2 |
21 |
2.12 |
0.03 |
7.3 |
52 |
-0.054 |
0.96 |
|
Table 3.14:
Analyse d'association des TagSNPs de CARD15 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
| |
|
Lot I |
| |
|
|
Stratifié selon: |
| |
|
|
|
présence HLA-Cw6 |
absence HLA-Cw6 |
| |
|
MAF |
P |
N |
Z |
P |
N |
| rs2076752 |
G>A |
33 |
0.005 |
58 |
-2.303 |
0.02 |
48 |
-1.514 |
0.13 |
| rs2111235 |
C>T |
29 |
0.40 |
59 |
-0.476 |
0.63 |
44 |
1.834 |
0.07 |
| rs8057341 |
G>A |
29.1 |
0.50 |
59 |
-0.626 |
0.53 |
44 |
1.801 |
0.07 |
| rs17221417 |
C>G |
35.5 |
0.01 |
58 |
-2.227 |
0.03 |
47 |
-1.040 |
0.30 |
| rs2066844 |
C>T |
7.4 |
0.04 |
17 |
-1.389 |
0.17 |
19 |
-1.461 |
0.14 |
| rs7203691 |
G>A |
36.2 |
0.11 |
69 |
2.436 |
0.01 |
51 |
-0.359 |
0.72 |
| rs10521209 |
T>G |
36.5 |
0.14 |
70 |
2.321 |
0.02 |
51 |
-0.508 |
0.61 |
| rs2066845 |
G>C |
1 |
NC |
5 |
*** |
*** |
2 |
*** |
*** |
| rs5743291 |
G>A |
10.9 |
0.12 |
28 |
2.939 |
0.003 |
26 |
-0.526 |
0.60 |
| rs1077861 |
A>T |
33.3 |
0.90 |
63 |
-1.145 |
0.25 |
46 |
1.586 |
0.11 |
| rs3135500 |
G>A |
37.3 |
0.29 |
67 |
2.546 |
0.01 |
55 |
-1.290 |
0.20 |
|
populations caucasiennes [Nair et al., 2001,Borgiani et al., 2002,Young et al., 2003,Plant et al., 2004,Lascorz et al., 2005]. Cependant, nos résultats semblent indiquer une contribution de ce gène dans la prédisposition au psoriasis. En effet, une preuve d'association, certes faible, entre la maladie et deux SNPs, rs2076752 dans la région 3'UTR et rs17221417 dans l'intron 2, indépendante du locus PSORS1, est observée après les différentes analyses sur nos deux cohortes françaises. De plus, dans nos 45 familles, il semble que le SNP rs5743291, (V955I) dans l'exon 9, soit associé à la maladie soit sous forme d'haplotype est conjointement avec l'allèle à risque HLA-Cw6. Lors de cette étude, nous nous sommes focalisés sur le gène lui-même en incorporant les informations venant de la littérature et des bases de données, parfois difficilement comparables entre elles. Une étude récente de la structure de DL de ce gène avec les données HapMap nous indique que les deux SNPs fortement associés dans notre étude sont en déséquilibre de liaison entre eux (r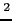 =0.89) et que rs17221417 est aussi en fort déséquilibre de liaison avec rs2066842 (P268S) dans l'exon 4 (r
=0.89) et que rs17221417 est aussi en fort déséquilibre de liaison avec rs2066842 (P268S) dans l'exon 4 (r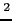 =0.96). Par conséquent, ce sont trois variants, un dans la région 5'UTR et deux codants non synonymes (V955I et P268S) qui pourraient intervenir sur la synthèse de la protéine normale. Le variant rs2066842 (P268S) provoque par exemple un changement d'acide aminé avec des propriétés chimiques différentes (hydrophobe vers hydrophile), pouvant modifier la forme finale de la protéine. Ce variant a été également montré comme étant associé à CD dans la population juive ashkénaze [Sugimura et al., 2003]. Cependant, lors de la seule autre étude globale du gène, aucune preuve d'association n'a été identifiée entre le psoriasis et les deux SNPs rs2066842 et rs2076752 [Plant et al., 2004]. Il existe d'autres variants codants non étudiés mais validés dans la population caucasienne.
Excepté des variants rares (<1%), la plupart sont des variants synonymes en fort déséquilibre de liaison avec un SNP déjà analysé dans notre population.
=0.96). Par conséquent, ce sont trois variants, un dans la région 5'UTR et deux codants non synonymes (V955I et P268S) qui pourraient intervenir sur la synthèse de la protéine normale. Le variant rs2066842 (P268S) provoque par exemple un changement d'acide aminé avec des propriétés chimiques différentes (hydrophobe vers hydrophile), pouvant modifier la forme finale de la protéine. Ce variant a été également montré comme étant associé à CD dans la population juive ashkénaze [Sugimura et al., 2003]. Cependant, lors de la seule autre étude globale du gène, aucune preuve d'association n'a été identifiée entre le psoriasis et les deux SNPs rs2066842 et rs2076752 [Plant et al., 2004]. Il existe d'autres variants codants non étudiés mais validés dans la population caucasienne.
Excepté des variants rares (<1%), la plupart sont des variants synonymes en fort déséquilibre de liaison avec un SNP déjà analysé dans notre population.
CARD15 semble être un facteur de prédisposition au psoriasis mais il serait nécessaire que ces résultats soient confirmés par d'autres études indépendantes (sur d'autres populations).




Next: Le gène CYLD (CYLinDromatosis
Up: Gènes «associés» au psoriasis
Previous: Exemple particulier du gène
Contents
anouar
2009-08-22
![]() B (Figure 3.5Fonction proposée de CARD15 dans la régulation de l'inflammation par l'intermédiaire de l'activation de la voie NF-$k$B en réponse à différents composants bactériens) avec la migration de NF-
B (Figure 3.5Fonction proposée de CARD15 dans la régulation de l'inflammation par l'intermédiaire de l'activation de la voie NF-$k$B en réponse à différents composants bactériens) avec la migration de NF-![]() B dans le noyau cellulaire et la transcription de nombreux gènes dont ceux codant des cytokines inflammatoires (de type 1), de chimiokines, responsables de l'inflammation. La protéine CARD15 pourrait donc intervenir dans la régulation de l'inflammation induite par les bactéries, c'est-à-dire, dans la protection de l'hôte contre les pathogènes. D'ailleurs, des études ont mis en évidence que la présence des 3 principales mutations de CARD15 observées dans la maladie de Crohn provoquait in vitro un défaut d'activation de la voie NF-
B dans le noyau cellulaire et la transcription de nombreux gènes dont ceux codant des cytokines inflammatoires (de type 1), de chimiokines, responsables de l'inflammation. La protéine CARD15 pourrait donc intervenir dans la régulation de l'inflammation induite par les bactéries, c'est-à-dire, dans la protection de l'hôte contre les pathogènes. D'ailleurs, des études ont mis en évidence que la présence des 3 principales mutations de CARD15 observées dans la maladie de Crohn provoquait in vitro un défaut d'activation de la voie NF-![]() B, partiel ou total, surtout en réponse à la stimulation par des agents bactériens (LPS ou PGN) [Ogura et al., 2001,Chamaillard et al., 2003]. De plus, une étude in vitro sur l'homologue murin de CARD15 (80% d'homologie en acide aminé), le gène Nod2, montre que deux
B, partiel ou total, surtout en réponse à la stimulation par des agents bactériens (LPS ou PGN) [Ogura et al., 2001,Chamaillard et al., 2003]. De plus, une étude in vitro sur l'homologue murin de CARD15 (80% d'homologie en acide aminé), le gène Nod2, montre que deux

