



Next: Protéines intervenant dans les
Up: Gènes «non associés» au
Previous: Les gènes codant des
Contents
Les gènes IL-1A, IL-1B, IL-1RN et IL-1R1
La cytokine pléiotrope, l'interleukine-1 (IL-1), est un médiateur important de l'inflammation et de l'immunité, particulièrement dans la peau [Camp et al., 1990,Mee et al., 2006]. Deux formes d'agoniste d'IL-1 (IL-1a et IL-1b) ont été identifiées ainsi qu'un antagoniste spécifique du récepteur IL-1 (IL-1ra/IL-1rn) et deux récepteurs de surface cellulaire identiques pour les deux formes d'agonistes d'IL-1, IL-1RI et IL-1RII, dont IL-1RII fonctionnant comme une molécule-leurre [Ravindran et al., 2004]. Les protéines d'IL-1 sont synthétisées par une large variété de types cellulaires comprenant les monocytes, les macrophages activés, les kératinocytes, les lymphocytes B stimulés, et les fibroblastes [Lord et al., 1991]. Leur sécrétion est stimulée entre autres par des produits bactériens, des toxines, des agressions physiques, et d'autres cytokines. L'IL-1 induit les effets systémiques de phase aiguë de l'inflammation tels que la fièvre. IL-1 augmente l'intensité de la réponse immune en stimulant entre autres la production de protéines nécessaires au cours de l'inflammation (prostaglandines, monoxyde d'azote etc..), et en particulier la synthèse des chémokines (IL-1, IL-8, IL-6 ou GM-CSF (granulocyte-macrophage colony-stimulating factor)). Elle stimule l'adhérence des leucocytes à l'endothélium en augmentant l'expression des molécules d'adhésion à la surface des cellules endothéliales (en particulier E-sélectine et ICAM-1)[Mee et al., 2006]. Elle favorise aussi la prolifération des fibroblastes, la synthèse de collagène et celle d'enzymes dégradant la matrice extracellulaire (collagénase). IL-1a et b se fixent sur les deux parties du récepteur de type I, IL-1RI, et entraînent une activation du domaine cytoplasmique TIR (analogue au récepteur Toll). Lors de l'activation, le récepteur de type I s'associe à une protéine accessoire (AcP) pour former un "hétérodimère" qui représente le récepteur complet. Le récepteur complet (récepteur de type I + AcP) recrute une kinase associée au récepteur de l'IL-1 (IRAK). La transduction du signal au noyau fait appel à une succession d'activation de kinases, en particulier celles impliquées dans les voies de signalisation NF-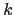 B et p38/MAPK, pour permettre la transcription de gènes cibles. IL-1ra ne se fixe que sur la partie IL-1RI et empêche l'activation du signal intracellulaire, provoquant ainsi le blocage des effets de l'IL-1. La fixation sur le récepteur de type II, IL-1RII, n'entraîne aucun signal intracellulaire. L'IL-1RI est retrouvé sur de très nombreuses cellules tandis qu'IL-1RII est exprimé principalement sur les neutrophiles, les monocytes et les lymphocytes B. Il existe aussi une forme soluble du récepteur de l'IL-1 de type II (sIL-1RII) qui fixe l'IL-1b avec une forte affinité, l'empêchant d'agir sur les cellules [Mee et al., 2006,Pietrzak et al., 2008].
B et p38/MAPK, pour permettre la transcription de gènes cibles. IL-1ra ne se fixe que sur la partie IL-1RI et empêche l'activation du signal intracellulaire, provoquant ainsi le blocage des effets de l'IL-1. La fixation sur le récepteur de type II, IL-1RII, n'entraîne aucun signal intracellulaire. L'IL-1RI est retrouvé sur de très nombreuses cellules tandis qu'IL-1RII est exprimé principalement sur les neutrophiles, les monocytes et les lymphocytes B. Il existe aussi une forme soluble du récepteur de l'IL-1 de type II (sIL-1RII) qui fixe l'IL-1b avec une forte affinité, l'empêchant d'agir sur les cellules [Mee et al., 2006,Pietrzak et al., 2008].
Des études ont montré que ces gènes du groupe d'IL-1 avaient un rôle dans plusieurs maladies inflammatoires. Les gènes IL1A et IL1B interviendraient dans la polyarthrite rhumatoïde [Cox et al., 1999,Kaijzel et al., 2002], la périodontite (inflammation des gencives) [Kornman et al., 1997,Diehl et al., 1999], l'ostéomyélite (inflammation de la moelle osseuse et du tissu osseux adjacent) [Asensi et al., 2003], la sclérose en plaques [Schrijver et al., 1999]. Le gène IL1RN aurait un rôle dans la polyarthrite rhumatoïde [Kaijzel et al., 2002], dans la sclérose en plaques [Schrijver et al., 1999] et dans le lupus érythémateux systémique [Blakemore et al., 1994]. L'ensemble de ces gènes, regroupés sur le chromosome 2q, pourrait donc avoir un rôle dans une autre maladie inflammatoire, le psoriasis.
De manière générale, le psoriasis est caractérisé par la dérégulation de cytokines, et en particulier la surexpression de cytokines pro-inflammatoires stimulant le recrutement des cellules du système immunitaire et la prolifération des kératinocytes (TNF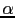 , IL-1, IL-8, IL-20 etc..). En vue de leur action sur différents types cellulaires impliqués dans cette maladie et de leur synthèse, en particulier dans les kératinocytes, les facteurs d'IL-1 ont été spécialement étudiés.
Contrairement aux attentes, lors des études in vivo sur les lésions d'individus atteints de psoriasis, l'activité fonctionnelle d'IL-1 est sensiblement réduite, comparée aux niveaux observées dans l'épiderme d'un individu sain et ceux dans la peau non-lésée d'un patient [Mee et al., 2006]. Ceci serait la conséquence d'une diminution d'IL-1a, combinée avec une augmentation d'IL-1b et d'inhibiteurs d'IL-1. Comparée aux biopsies de tissus sains, une augmentation nette du rapport d'IL-1ra sur IL-1a, ainsi qu'une induction d'IL-1RII, est constatée dans les lésions [Reich et al., 2002,Mee et al., 2006]. Une étude transcriptionnelle de l'expression des gènes qui compare des biopsies de peau lésée et non-lésée obtenues chez les mêmes patients atteints de psoriasis, montre des données comparables [Mee et al., 2006]. Une diminution des niveaux d'ARNm de l'IL-1a ainsi qu'une augmentation nette de l'ARNm d'IL-1b sont observées dans les biopsies de la peau lésée. Aucune variation d'ARNm entre les deux types de biopsies n'a été observée pour l'antagoniste du récepteur IL-1 (IL-1ra), ni d'ailleurs pour les deux transcripts du récepteur IL-1. La présence de niveaux élevés de protéines IL-1RII, malgré le manque d'induction d'ARNm IL-1RII dans l'épiderme lésé, peut résulter de mécanismes post-transcriptionnels [Mee et al., 2006]. Cette étude montre une augmentation de l'activité générale d'IL-1, déduite par l'augmentation observée dans l'épiderme lésé de l'expression d'une cytokine secondaire, connue pour être induite par IL-1, l'IL-8. Des expériences de génomique fonctionnelle, comparant des profils de transcriptome dérivés de la peau lésée de psoriasis et des kératinocytes stimulés par IL-1a, ont démontré un niveau élevé de similitudes entre les deux profils dont ceux des gènes codant pour le complexe de différenciation épidermique et pour des molécules antimicrobiennes qui sont hautement surexprimés [Mee et al., 2006].
Des études, sur des souris transgéniques sur-exprimant constitutivement les molécules de la famille IL-1 dans l'épiderme basal, montrent que la surexpression d'IL-1a est suffisante pour enclencher l'inflammation cutanée spontanément avec des similitudes histologiques des lésions de psoriasis [Groves et al., 1995]. De plus, le phénotype est aggravé chez les souris transgéniques sur-exprimant IL-1a et IL-1RI [Groves et al., 1996]. La surexpression d'IL-1RII dans le même système a comme conséquence la sous-régulation spécifique de l'inflammation cutanée médiée par l'IL-1 [Rauschmayr et al., 1997]. Une inflammation cutanée psoriasiforme ainsi qu'une arthrite inflammatoire par l'intermédiaire du facteur TNF
, IL-1, IL-8, IL-20 etc..). En vue de leur action sur différents types cellulaires impliqués dans cette maladie et de leur synthèse, en particulier dans les kératinocytes, les facteurs d'IL-1 ont été spécialement étudiés.
Contrairement aux attentes, lors des études in vivo sur les lésions d'individus atteints de psoriasis, l'activité fonctionnelle d'IL-1 est sensiblement réduite, comparée aux niveaux observées dans l'épiderme d'un individu sain et ceux dans la peau non-lésée d'un patient [Mee et al., 2006]. Ceci serait la conséquence d'une diminution d'IL-1a, combinée avec une augmentation d'IL-1b et d'inhibiteurs d'IL-1. Comparée aux biopsies de tissus sains, une augmentation nette du rapport d'IL-1ra sur IL-1a, ainsi qu'une induction d'IL-1RII, est constatée dans les lésions [Reich et al., 2002,Mee et al., 2006]. Une étude transcriptionnelle de l'expression des gènes qui compare des biopsies de peau lésée et non-lésée obtenues chez les mêmes patients atteints de psoriasis, montre des données comparables [Mee et al., 2006]. Une diminution des niveaux d'ARNm de l'IL-1a ainsi qu'une augmentation nette de l'ARNm d'IL-1b sont observées dans les biopsies de la peau lésée. Aucune variation d'ARNm entre les deux types de biopsies n'a été observée pour l'antagoniste du récepteur IL-1 (IL-1ra), ni d'ailleurs pour les deux transcripts du récepteur IL-1. La présence de niveaux élevés de protéines IL-1RII, malgré le manque d'induction d'ARNm IL-1RII dans l'épiderme lésé, peut résulter de mécanismes post-transcriptionnels [Mee et al., 2006]. Cette étude montre une augmentation de l'activité générale d'IL-1, déduite par l'augmentation observée dans l'épiderme lésé de l'expression d'une cytokine secondaire, connue pour être induite par IL-1, l'IL-8. Des expériences de génomique fonctionnelle, comparant des profils de transcriptome dérivés de la peau lésée de psoriasis et des kératinocytes stimulés par IL-1a, ont démontré un niveau élevé de similitudes entre les deux profils dont ceux des gènes codant pour le complexe de différenciation épidermique et pour des molécules antimicrobiennes qui sont hautement surexprimés [Mee et al., 2006].
Des études, sur des souris transgéniques sur-exprimant constitutivement les molécules de la famille IL-1 dans l'épiderme basal, montrent que la surexpression d'IL-1a est suffisante pour enclencher l'inflammation cutanée spontanément avec des similitudes histologiques des lésions de psoriasis [Groves et al., 1995]. De plus, le phénotype est aggravé chez les souris transgéniques sur-exprimant IL-1a et IL-1RI [Groves et al., 1996]. La surexpression d'IL-1RII dans le même système a comme conséquence la sous-régulation spécifique de l'inflammation cutanée médiée par l'IL-1 [Rauschmayr et al., 1997]. Une inflammation cutanée psoriasiforme ainsi qu'une arthrite inflammatoire par l'intermédiaire du facteur TNF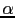 sont observées chez les souris où le gène IL-1ra est détruit [Horai et al., 2004,Shepherd et al., 2004].
sont observées chez les souris où le gène IL-1ra est détruit [Horai et al., 2004,Shepherd et al., 2004].
Bien que la plupart des observations de la peau lésée suggèrent une sous-régulation de l'activité d'IL-1 contrairement aux observations faites chez les modèles murins, il est important de noter que cette réponse à IL-1 varie en fonction des concentrations de ligands extracellulaires biologiquement actifs (IL-1a et IL-1b), d'IL-1ra et des niveaux d'expression des deux types de récepteur IL-1 (IL-1RI et IL-1RII). Ainsi, l'équilibre entre les agonistes (IL-1a/b et IL-1RI) et les antagonistes (IL-1ra et IL-1RII) du système IL-1 peut jouer un rôle principal dans le processus d'inflammation cutanée [Mee et al., 2006].
Des études d'association ont confirmé le rôle des protéines du système IL-1 dans la maladie. Une première étude anglaise montre une association entre un polymorphisme d'IL-1ra et le psoriasis à début précoce [Tarlow et al., 1997]. Lors d'une étude allemande cas-témoins (231 versus 345) sur l'implication de polymorphismes d'IL-1b (C-511T, T+3953C) et d'IL-1ra (intron 2) dans le psoriasis, le polymorphisme C-511T d'IL-1b est montré associé à une production élevée d'IL-1ra en réponse à des lipopolysaccharides et de l'IL-10 et au psoriasis à début tardif (P=0.04) [Reich et al., 2002]. Néanmoins, une étude japonaise cas-témoins (170 versus 210) ne révèle aucune association entre le psoriasis et IL-1a, IL-1b et IL1-ra [Chang et al., 2007]. De plus, d'autres équipes ont testé ces gènes pour l'arthrite psoriasique (PsA). Dans une étude d'association cas-témoins (140 versus 100 caucasiens) sur les polymorphismes d'IL-1b (T+3953C), d'IL-1a (C-889T) et IL-1R1 (C+970T), seule une association entre le PsA et le polymorphisme d'IL-1a a été observée (P=0.02) [Ravindran et al., 2004]. D'autres études ont confirmé ces résultats. L'absence d'association entre PsA et IL-1b ou d'IL-1ra est aussi observée dans une étude irlandaise cas-témoins (147 versus 389) [Balding et al., 2003]. Une étude supplémentaire canadienne (220 versus 150) confirme l'association du gène IL-1a dans le PsA [Rahman et al., 2006]. Néanmoins, ce gène ne semble pas intervenir dans la population japonaise (170 versus 210) et dans la population de l'île de Terre Neuve (226 versus 95) puisqu'aucune association n'a été identifiée entre le PsA et IL-1a, IL-1b et IL1-ra [Peddle et al., 2005,Chang et al., 2007].
L'ensemble de ces données suggère que toutes les molécules du système IL-1 sont susceptibles d'être des médiateurs importants dans le déclenchement et l'entretien des plaques de psoriasis et peuvent représenter des gènes candidats. Nous avons donc réalisé une étude d'association globale de ces gènes (IL-1A, IL-1B, IL-RA et IL1RN) dans notre population française. Avec la même méthode que lors des études sur les gènes précédents, nous avons sélectionné 35 SNPs, choisis en fonction du déséquilibre de liaison existant entre les SNPs validés dans les bases de données (HapMap/NCBI) présents à l'intérieur des gènes (Figures 3.9Structure DL du gène IL1R1, 3.10Structure DL du gène IL1A et 3.11Structure DL du gène IL1RN). L'ensemble des renseignements sur les SNPs sélectionnés ainsi que les résultats obtenus lors de leurs analyses dans le lot I par le programme FBAT individuellement ou conjointement sont décrits dans les tableaux 3.31Identification et analyse d'association des TagSNPs de IL1R1, IL1A, IL1B et IL1RN sur le Lot I par FBAT et 3.32 Analyse d'association des haplotypes de IL1R1, IL1A, IL1B et IL1RN sur le Lot I par FBAT. L'analyse des TagSNPs lors de la stratification des patients du lot I selon la présence ou non de l'haplotype à risque révèle seulement une association faible entre le variant d'IL-1B (rs3917354) et le psoriasis (P=0.03) lors de l'analyse des individus non porteurs d'HLA-Cw6 (Table 3.33Analyse d'association des TagSNPs dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I). Il ne semble donc pas exister une interaction entre ces deux gènes dans la pathogenèse du psoriasis.
L'ensemble de ces analyses nous indique qu'aucun des gènes ne contribue significativement à la pathogenèse du psoriasis, confirmant les études précédemment détaillées [Reich et al., 2002,Chang et al., 2007]. Néanmoins, l'étude allemande observe une association entre le gène IL-1B et le psoriasis à début tardif [Reich et al., 2002]. Et dans notre étude, une association faible a été obtenue lorsque les individus non porteurs du HLA-Cw6 seulement du lot I sont analysés. De nombreuses études ont mis en évidence que contrairement aux patients du type I (début précoce), les patients de type II (début tardif) étaient peu porteurs de l'haplotype à risque, le HLA-Cw6. Or, la grande majorité de nos patients du premier lot sont de type I et porteur du HLA-Cw6, ce qui peut peut être expliquer la faible association observée. Cette hypothèse reste néanmoins à être confirmée.
Le gène IL12RB1
Un déséquilibre des réponses Th1/Th2 au profit des cytokines pro-inflammatoires de type 1 (IL-2, IFN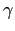 , TNF
, TNF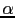 et IL-17) est observé dans le psoriasis. Une cytokine clé pour le développement des réponses Th1 est l'interleukine-12 (IL-12). IL-12, produit par les monocytes et les cellules présentatrices d'antigène, joue en effet
et IL-17) est observé dans le psoriasis. Une cytokine clé pour le développement des réponses Th1 est l'interleukine-12 (IL-12). IL-12, produit par les monocytes et les cellules présentatrices d'antigène, joue en effet
Figure 3.9:
Structure DL du gène IL1R1
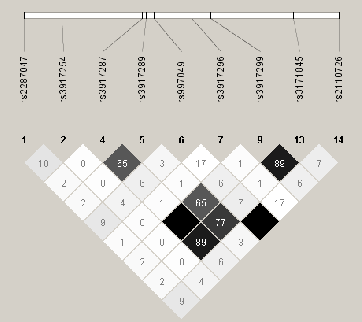 |
Figure 3.10:
Structure DL du gène IL1A
 |
Figure 3.11:
Structure DL du gène IL1RN
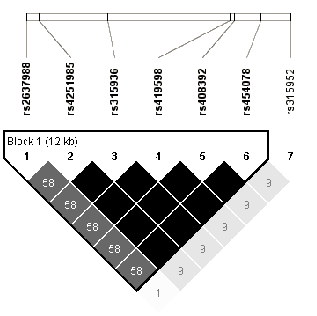 |
Table 3.31:
Identification et analyse d'association des TagSNPs de IL1R1, IL1A, IL1B et IL1RN sur le Lot I par FBAT
| |
|
|
|
|
|
|
|
FBAT
Lot I |
| |
|
|
|
| |
rs2192752 |
|
|
4838958 |
A>C |
25 |
D |
22.9 |
83 |
-0.843 |
0.40 |
| |
rs3917335 |
Intron 1 |
|
4840878 |
C>T |
5.9 |
D |
4.3 |
17 |
0.043 |
| |
rs871657 |
Intron 1 |
|
4840926 |
C>T |
14.2 |
D |
19.7 |
80 |
1.233 |
| |
rs3917238 |
Intron 1 |
|
4842668 |
C>T |
38.1 |
D |
|
|
|
| |
rs3917254 |
Intron 2 |
|
4846103 |
G>A |
5.8 |
H |
9.6 |
35 |
0.234 |
| |
rs3917273 |
Intron 2 |
|
4849304 |
A>T |
37.3 |
D |
|
|
|
| |
rs3917289 |
Intron 4 |
|
4851496 |
G>T |
8.3 |
H |
6.9 |
41 |
-0.769 |
| |
rs997049 |
Intron 4 |
|
4852018 |
A>T |
43.3 |
H |
|
|
|
| |
rs3917296 |
Intron 5 |
|
4854418 |
A>G |
11.7 |
H |
11.5 |
37 |
-0.346 |
| |
rs3917299 |
Intron 6 |
|
4855671 |
A>G |
7.5 |
H |
5.9 |
36 |
-0.330 |
| |
rs4848300 |
|
|
2235829 |
T>C |
30.8 |
D |
20.7 |
91 |
0.319 |
0.75 |
| |
rs3783550 |
Intron 6 |
|
2240808 |
A>C |
29.2 |
D |
30.1 |
99 |
-0.017 |
| |
rs3783548 |
Intron 6 |
|
2241256 |
T>C |
7 |
D |
|
|
|
| |
rs3783546 |
Intron 6 |
|
2242753 |
G>C |
29.2 |
H |
30.1 |
99 |
-0.017 |
| |
rs3783539 |
Intron 4 |
|
2245502 |
G>A |
23.7 |
D |
30.1 |
99 |
0.005 |
| |
rs2856838 |
Intron 3 |
|
2247895 |
C>T |
40 |
D |
49.3 |
101 |
-0.124 |
| |
rs1800587 |
5' UTR |
|
2250883 |
C>T |
30.8 |
A |
21.7 |
86 |
0.404 |
| |
rs1143643 |
Intron 6 |
|
2296225 |
G>A |
39.2 |
H |
36.8 |
99 |
0.687 |
0.49 |
| |
rs1143634 |
Exon 5 |
F105F |
2298313 |
C>T |
22.3 |
D |
18.9 |
76 |
-0.668 |
| |
rs1143633 |
Intron 4 |
|
2298390 |
G>A |
39.7 |
D |
36 |
100 |
0.423 |
| |
rs3136558 |
Intron 3 |
|
2299198 |
T>C |
30.4 |
D |
18.1 |
72 |
-1.143 |
| |
rs3136557 |
Intron 3 |
|
2299265 |
C>T |
21.7 |
D |
|
|
|
| |
rs3917356 |
Intron 3 |
|
2300286 |
G>A |
45 |
D |
|
|
|
| |
rs3917354 |
Intron 3 |
|
2300818 |
T>- |
23.9 |
D |
15.5 |
65 |
-1.068 |
| |
rs1143629 |
Intron 2 |
|
2301441 |
T>C |
16.7 |
D |
35.1 |
98 |
-0.421 |
| |
rs2592346 |
Intron 1 |
|
2584946 |
T>C |
25 |
D |
41.1 |
101 |
0.517 |
0.60 |
| |
rs928940 |
Intron 1 |
|
2585418 |
T>G |
11.7 |
D |
13.6 |
56 |
0.536 |
| |
rs878972 |
Intron 1 |
|
2585636 |
A>C |
17.4 |
D |
26.6 |
89 |
0.167 |
| |
rs315934 |
Intron 1 |
|
2591629 |
A>G |
28.3 |
D |
19.7 |
61 |
-0.973 |
| |
rs3087262 |
Intron 1 |
|
2592125 |
G>C |
8.7 |
D |
14.3 |
56 |
0.220 |
| |
rs1794067 |
Intron 2 |
|
2594307 |
G>A |
27.1 |
D |
27.2 |
92 |
0.108 |
| |
rs2232354 |
Intron 3 |
|
2595258 |
T>G |
28.6 |
D |
|
|
|
| |
rs579543 |
Intron 4 |
|
2597554 |
C>T |
27.1 |
D |
28.3 |
92 |
0.415 |
| |
rs315952 |
Exon 5 |
S112S |
2598227 |
T>C |
25.8 |
H |
27.8 |
80 |
-0.340 |
| |
rs4252041 |
3' UTR |
|
2598533 |
C>T |
6.8 |
D |
2.4 |
17 |
-0.983 |
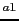 associé à la spondylarthrite ankylosante (Wu and Gu, 2007), associé à la spondylarthrite ankylosante (Wu and Gu, 2007), 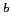 NM_000877 pour IL1R1, NM_000575 pour IL1A, NM_000576 pour IL1B, NM_000577 pour IL1RN, NM_000877 pour IL1R1, NM_000575 pour IL1A, NM_000576 pour IL1B, NM_000577 pour IL1RN, 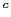 NT_022171.14 pour IL1R1, NT_022135.15 pour IL1A, IL1B et IL1RN NT_022171.14 pour IL1R1, NT_022135.15 pour IL1A, IL1B et IL1RN |
|
|
Table 3.32:
Analyse d'association des haplotypes de IL1R1, IL1A, IL1B et IL1RN sur le Lot I par FBAT
|
|
Lot I |
|
|
|
|
MAF |
|
|
H1 |
ACCGGAA |
34.4 |
0.89 |
|
|
H2 |
ACTGGAA |
19.9 |
|
|
H3 |
CCCGGAA |
19.5 |
|
|
H4 |
ACCGGGA |
6.9 |
|
|
H5 |
ACCAGAA |
6.2 |
|
|
H6 |
ACCGTAG |
5.9 |
|
|
H1 |
TAGGTC |
43 |
0.82 |
|
|
H2 |
TCCACC |
33.1 |
|
|
H3 |
CAGGCT |
23.7 |
|
|
H1 |
ACATTT |
30.7 |
0.23 |
|
|
H2 |
GCGTTC |
30 |
|
|
H3 |
GTGC-T |
15 |
|
|
H4 |
GCGTTT |
12.5 |
|
|
H1 |
TTAAGGCTC |
37.1 |
0.53 |
|
|
H2 |
CTCAGATTC |
25.3 |
|
|
H3 |
CGAACGCCC |
11.9 |
|
|
H4 |
TTAGGGCCC |
10.6 |
|
|
H5 |
TTAGGGCTC |
5.1 |
Table 3.33:
Analyse d'association des TagSNPs dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I
|
|
|
Lot I |
|
|
|
|
|
Stratifié selon: |
|
|
|
|
|
|
présence HLA-Cw6 |
|
|
|
|
MAF |
P |
N |
Z |
P |
|
|
rs2192752 |
A>C |
22.9 |
0.40 |
53 |
0.669 |
0.50 |
43 |
-2.009 |
0.05 |
|
|
rs3917335 |
C>T |
4.3 |
0.97 |
9 |
*** |
*** |
11 |
0.205 |
|
|
rs871657 |
C>T |
19.7 |
0.22 |
50 |
0.694 |
0.49 |
45 |
1.045 |
|
|
rs3917254 |
G>A |
9.6 |
0.82 |
22 |
0.577 |
0.56 |
19 |
-0.310 |
|
|
rs3917289 |
G>T |
6.9 |
0.44 |
28 |
-0.413 |
0.68 |
20 |
-0.640 |
|
|
rs3917296 |
A>G |
11.5 |
0.73 |
23 |
-1.424 |
0.15 |
21 |
1.059 |
|
|
rs3917299 |
A>G |
5.9 |
0.74 |
27 |
-0.152 |
0.88 |
16 |
-0.310 |
|
|
rs4848300 |
T>C |
20.7 |
0.75 |
57 |
0.510 |
0.61 |
49 |
-0.134 |
0.89 |
|
|
rs3783550 |
A>C |
30.1 |
0.99 |
58 |
-1.094 |
0.27 |
52 |
1.125 |
|
|
rs3783546 |
G>C |
30.1 |
0.99 |
58 |
-1.094 |
0.27 |
52 |
1.125 |
|
|
rs3783539 |
G>A |
30.1 |
0.99 |
58 |
-1.094 |
0.27 |
52 |
1.159 |
|
|
rs2856838 |
C>T |
49.3 |
0.90 |
63 |
0.386 |
0.70 |
51 |
-0.599 |
|
|
rs1800587 |
C>T |
21.7 |
0.69 |
54 |
1.047 |
0.30 |
46 |
-0.629 |
|
|
rs1143643 |
G>A |
36.8 |
0.49 |
59 |
0.370 |
0.71 |
53 |
0.584 |
0.56 |
|
|
rs1143634 |
C>T |
18.9 |
0.50 |
46 |
0.534 |
0.59 |
43 |
-1.454 |
|
|
rs1143633 |
G>A |
36 |
0.67 |
60 |
0.353 |
0.72 |
54 |
0.224 |
|
|
rs3136558 |
T>C |
18.1 |
0.25 |
41 |
-0.079 |
0.94 |
44 |
-1.436 |
|
|
rs3917354 |
T>- |
15.5 |
0.29 |
35 |
0.714 |
0.48 |
41 |
-2.188 |
|
|
rs1143629 |
T>C |
35.1 |
0.67 |
58 |
-1.476 |
0.14 |
51 |
1.079 |
|
|
rs2592346 |
T>C |
41.1 |
0.60 |
62 |
0.690 |
0.49 |
52 |
-0.005 |
0.99 |
|
|
rs928940 |
T>G |
13.6 |
0.59 |
32 |
0.457 |
0.65 |
30 |
0.284 |
|
|
rs878972 |
A>C |
26.6 |
0.87 |
59 |
-0.141 |
0.89 |
44 |
0.417 |
|
|
rs315934 |
A>G |
19.7 |
0.33 |
40 |
-1.932 |
0.05 |
30 |
0.814 |
|
|
rs3087262 |
G>C |
14.3 |
0.83 |
33 |
0.660 |
0.51 |
29 |
-0.390 |
|
|
rs1794067 |
G>A |
27.2 |
0.92 |
61 |
-0.242 |
0.81 |
45 |
0.449 |
|
|
rs579543 |
C>T |
28.3 |
0.68 |
58 |
0.004 |
0.99 |
48 |
0.591 |
|
|
rs315952 |
T>C |
27.8 |
0.73 |
49 |
-0.669 |
0.50 |
41 |
0.230 |
|
|
rs4252041 |
C>T |
2.4 |
0.33 |
14 |
0.480 |
0.63 |
7 |
*** |
un rôle important dans la régulation de la différenciation des cellules T CD4+ naïves en cellules Th1 et favorise l'immunité à médiation cellulaire ainsi que l'induction de la production d'IFN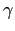 par les cellules T [Watford et al., 2003]. Ce dernier facilite l'infiltration et l'activation des cellules T dans l'épiderme par l'augmentation de l'expression de molécules d'adhésion sur les leucocytes, les cellules endothéliales et les kératinocytes (ICAM-1 et HLA-DR).
par les cellules T [Watford et al., 2003]. Ce dernier facilite l'infiltration et l'activation des cellules T dans l'épiderme par l'augmentation de l'expression de molécules d'adhésion sur les leucocytes, les cellules endothéliales et les kératinocytes (ICAM-1 et HLA-DR).
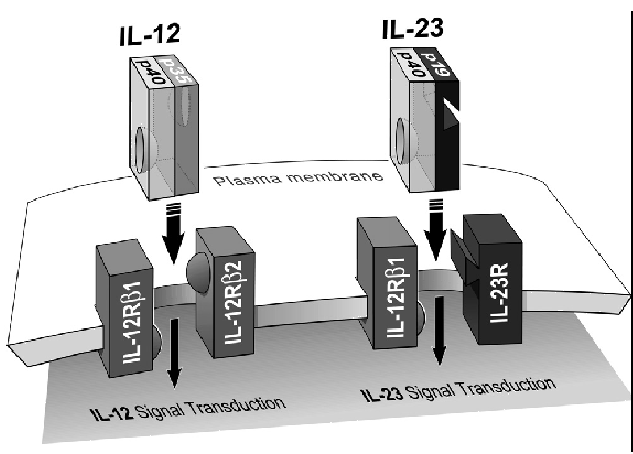
IL-12 est une cytokine hétéro-dimérique composée de deux sous-unités (p35 et p40) (Figure 3.12Représentation schématique de la structure des cytokines IL-12 et IL-23 et de leurs récepteurs).
Cette cytokine transmet son signal en se liant à son récepteur (IL-12R), constitué de deux sous-unités IL-12R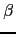 1 et IL-12R
1 et IL-12R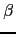 2, qui est exprimé sur les cellules T et NK activés [Presky et al., 1996]. Une étude montre que la haute affinité d'IL-12R au ligand IL-12 nécessite la co-expression de ces deux sous unités, confirmée par une étude sur des souris déficientes en IL-12R [Wu et al., 1997,Wu et al., 2000]. Une autre cytokine, l'IL-23, partage une sous-unité avec l'IL-12, IL-12p40. Le récepteur d'IL-23 partage une sous-unité commune à IL-12R, IL-12 R
2, qui est exprimé sur les cellules T et NK activés [Presky et al., 1996]. Une étude montre que la haute affinité d'IL-12R au ligand IL-12 nécessite la co-expression de ces deux sous unités, confirmée par une étude sur des souris déficientes en IL-12R [Wu et al., 1997,Wu et al., 2000]. Une autre cytokine, l'IL-23, partage une sous-unité avec l'IL-12, IL-12p40. Le récepteur d'IL-23 partage une sous-unité commune à IL-12R, IL-12 R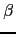 1, sur laquelle la partie IL-12p40 se lie (Figure 3.12Représentation schématique de la structure des cytokines IL-12 et IL-23 et de leurs récepteurs). La cytokine pro-inflammatoire IL-23, produite par les macrophages et les cellules dendritiques, active les cellules T, qui produisent l'IL-17, et induit la production de cytokines par les cellules endothéliales, les kératinocytes et les macrophages [Torti and Feldman, 2007].
1, sur laquelle la partie IL-12p40 se lie (Figure 3.12Représentation schématique de la structure des cytokines IL-12 et IL-23 et de leurs récepteurs). La cytokine pro-inflammatoire IL-23, produite par les macrophages et les cellules dendritiques, active les cellules T, qui produisent l'IL-17, et induit la production de cytokines par les cellules endothéliales, les kératinocytes et les macrophages [Torti and Feldman, 2007].
De nombreuses études soutiennent une implication des deux cytokines, IL-12 et IL-23, dans la pathogenèse du psoriasis. Par exemple, une étude montre une expression élevée de l'IL-23p19 (la sous-unité spécifique d'IL-23) et de p40 (la sous-unité commune à IL-12 et IL-23) dans les lésions psoriasiques comparées à la peau non lésée de patients ou à la peau saine [Chen et al., 2006]. En revanche, aucune différence d'expression n'a été trouvée pour la sous-unité p35 (sous-unité spécifique d'IL-12) dans les différentes peaux étudiées, indiquant ainsi un rôle important de la sous-unité commune de l'IL-12 et IL-23, p40, dans la pathogenèse du psoriasis [Chen et al., 2006,Shaker et al., 2006]. L'expression diminuée de ces cytokines lors de différents traitements du psoriasis (UVA, cyclosporine, etanercept, alefacept) et le bon fonctionnement des nouvelles thérapies ciblant cette sous unité p40 confirment le rôle important de ces molécules dans la génèse de la maladie [Torti and Feldman, 2007]. Une étude japonaise cas-témoins (143 cas versus 100 témoins) a identifié une association entre le psoriasis et un SNP (rs3212227) présent dans la région 3'UTR du gène IL12B codant la sous-unité p40 (commune aux deux interleukines) (P=0.035) [Tsunemi et al., 2002]. Quatre autres études réalisées sur d'autres populations (Angleterre (318 vs 288), USA (1446 vs 1432), Chine (170 vs 210) et caucasien (1810 vs 2522)) confirment d'ailleurs une association significative de ce gène avec le psoriasis ((0.0001<P<0.04), (P=7.85x10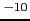 ), (P=0.0067) (P=1.7x10
), (P=0.0067) (P=1.7x10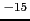 ) respectivement) [Capon et al., 2007,Cargill et al., 2007,Chang et al., 2007,Nair et al., 2008]. Le variant en 3'UTR du gène a aussi
) respectivement) [Capon et al., 2007,Cargill et al., 2007,Chang et al., 2007,Nair et al., 2008]. Le variant en 3'UTR du gène a aussi
Figure 3.12:
Représentation schématique de la structure des cytokines IL-12 et IL-23 et de leurs récepteurs
Les deux cytokines se composent d'une même sous-unité, p40 qui se lie à une sous-unité commune aux deux récepteurs, IL-12RB1. (D'après [Torti and Feldman, 2007])
 |
été associé avec d'autres maladies auto-immunes comme le diabète de type I, la dermatite atopique et l'asthme [Morahan et al., 2001,Tsunemi et al., 2002,Randolph et al., 2004]. De plus, dans une étude japonaise cas-témoins (382 AD versus 658), deux polymorphismes (rs436857, rs393548) situés sur le chromosome 19p13, au niveau du promoteur du gène codant la sous-unité commune aux récepteurs de l'IL-12 et l'IL-23 (IL-12 R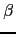 1) sont montrés associés avec l'AD (P=0.00044/0.00075) [Takahashi et al., 2005].
1) sont montrés associés avec l'AD (P=0.00044/0.00075) [Takahashi et al., 2005].
Toutes ces observations suggèrent l'implication de ces cytokines ainsi que de leurs récepteurs dans le psoriasis mais aussi dans différentes maladies inflammatoires. La particularité de la sous-unité IL-12 R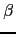 1, d'être commune aux récepteurs de deux interleukines d'intérêts (l'IL-12 et l'IL-23) ainsi que la présence du gène codant cette sous-unité dans un des locus de prédisposition au psoriasis sur le chromosome 19p13 (PSORS6) suggèrent le gène IL-12RB1 comme un gène candidat.
1, d'être commune aux récepteurs de deux interleukines d'intérêts (l'IL-12 et l'IL-23) ainsi que la présence du gène codant cette sous-unité dans un des locus de prédisposition au psoriasis sur le chromosome 19p13 (PSORS6) suggèrent le gène IL-12RB1 comme un gène candidat.
Nous avons analysé huit TagSNPs communs préalablement identifiés lors de l'étude de ce gène dans la prédisposition à la dermatite atopique dans une population japonaise [Takahashi et al., 2005]. Les résultats obtenus lors de l'analyse individuelle ou conjointe de ces variants par FBAT sur nos 45 familles sont présentés dans le tableau 3.34Identification et analyse d'association des TagSNPs de IL12RB1 sur le Lot I par FBAT et 3.35 Analyse d'association des haplotypes de IL12RB1 sur le Lot I par FBAT. Aucune association entre le psoriasis et le gène IL-12 RB1 n'a été observée. La stratification des 45 familles selon l'haplotype à risque en PSORS1 ne permet pas non plus de montrer une association entre le gène et le psoriasis (Tableau 3.36Analyse d'association des TagSNPs de IL12RB1 dans les sous groupes porteurs ou non de l'allèle à risque HLA-Cw6 dans le Lot I). Cependant, nous ne pouvons pas exclure qu'étudier seulement les TagSNPs représentatifs des haplotypes présents dans la population japonaise puissent fausser notre étude faite sur une population française. Mais, cela semble peu probable car excepté pour un variant (rs11086087), les fréquences alléliques de ces variants sont assez comparables dans les deux types de populations (Tableau 3.34Identification et analyse d'association des TagSNPs de IL12RB1 sur le Lot I par FBAT).
De plus, le manque d'association avec le gène codant une partie du récepteur d'IL-12 et d'IL-23 semble être en accord avec deux autres grandes études anglaises et américaines [Capon et al., 2007,Cargill et al., 2007].
En conclusion, le gène IL-12 RB1 ne semble pas jouer un rôle dans la pathogenèse du psoriasis dans notre échantillon.




Next: Protéines intervenant dans les
Up: Gènes «non associés» au
Previous: Les gènes codant des
Contents
anouar
2009-08-22



