



Next: Génotypage par la méthode
Up: Technique
Previous: Technique
Contents
Le choix des couples d'amorces a été réalisé avec difficulté, dû à la grande variabilité connue de cette région. Nous avons donc sélectionné, après l'étude de la structure des introns et des exons du gène HLA-C, les parties peu polymorphes du gène de façon à pouvoir séquencer l'intégralité de la région. La sélection précise des deux couples d'amorces pour séquencer l'exon 2 et 3 a été réalisée par le programme "Primer 3" (http://www-genome.wi.mit.edu/genome_software/other/ primer3.html) en tenant compte de certains critères comme la température d'hybridation optimale de 60 c (avec la formule 4GC+2AT- 4) et la taille de l'amorce d'une vingtaine de nucléotides (20 mers) [Rozen and Skaletsky, 2000](Tableau 3.2.1Contexte sur le locus HLA-C).
Table 2.6:
Conditions choisies pour l'amplification des exons 2 et 3 du gène HLA-C
| |
3cmTaille du fragment (en pb) |
3cmTempérature d'hybridation (enc) |
| HLA-C_exon2 |
411 |
57 |
| HLA-C_exon3 |
488 |
55 |
Table 2.7:
Mélange réactionnel pour une amplification individuelle des exons 2 et 3 par PCR sur une plaque de 96 puits
(pour l'exon 3 : une deuxième amplification était souvent nécessaire)
| Produits |
Quantité (en  ) ) |
ADNg non amplifié à 5ng/ |
5 |
| Tampon 10X |
2.5 |
Amorces 2F à 10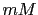 |
0.5 |
Amorces 2R à 10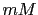 |
0.5 |
dNTP à 2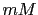 |
2.5 |
| Eau |
12.6 |
MgCl2 à 25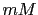 |
1.3 |
| Enzyme Taq GOLD |
0.1 |
| Volume final |
25 |
Table 2.8:
Différentes étapes du cycle de la PCR, variant selon la température d'hybridation des amorces pour chaque exon
| Température en c |
Temps |
Cycles |
| 96 |
5 min |
X1 |
| 94 |
40 sec. |
|
| 57/55 |
40 sec. |
| 72 |
1 min. |
| 72 |
5 min. |
X1 |
| 15 |
 |
|
Les couples d'amorces définis par cet outil sont ensuite testés, grâce à un gradient de température, sur un témoin "test" provenant de familles du CEPH, afin de vérifier si l'hybridation entre les amorces et l'ADN est spécifique et quelle est la température optimale. L'analyse des produits amplifiés déposés sur gel d'agarose à 2% permet de vérifier la taille des fragments obtenus et de contrôler l'amplification, en comparaison avec un témoin négatif (l'eau à la place de l'ADN) et un marqueur de taille (MXIV). S'il n'y a pas eu d'amplification ou amplification non spécifique, les amorces seront remplacées par d'autres plus performantes. Le protocole optimisé pour chaque exon est décrit dans les tableaux 2.6Conditions choisies pour l'amplification des exons 2 et 3 du gène HLA-C, 2.7Mélange réactionnel pour une amplification individuelle des exons 2 et 3 par PCR sur une plaque de 96 puits et 2.8Différentes étapes du cycle de la PCR, variant selon la température d'hybridation des amorces pour chaque exon. Après contrôle de l'amplification de l'ADN par dépôt sur gel d'agarose à 2%, une purification des produits de PCR doit être effectuée. Cette étape s'effectue en déposant chaque produit de PCR sur une colonne contenant du Bio-Gel P-100 Gel (Bio-Rad) et en les centrifugeant 4 minutes à 500g. Ceci permet d'obtenir une solution dépourvue de toute impureté comme les amorces, les dNTPs, l'enzyme. On obtient ainsi de l'ADN amplifié et purifié. La réaction de séquençage est effectuée selon le type de séquenceur utilisé dans un volume final de 15 (10
(10 dans le cas du séquenceur 3730) à partir de 1 à 2 ou 3
dans le cas du séquenceur 3730) à partir de 1 à 2 ou 3 de produit de PCR purifié (selon les résultats quantitatifs de l'amplification), 1
de produit de PCR purifié (selon les résultats quantitatifs de l'amplification), 1 de l'amorce sens (5'->3') ou antisens (3'->5') à 10
de l'amorce sens (5'->3') ou antisens (3'->5') à 10 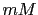 , selon le séquençage dans les sens 5'->3' ou le sens 3'->5', et 2
, selon le séquençage dans les sens 5'->3' ou le sens 3'->5', et 2 (1
(1 ) d'un mélange réactionnel de séquençage, le Big Dye (PE Biosystems, ABI prism). Il contient les ddNTPs, les dNTPs, le MgCl2, le tampon et la Taq polymérase. L'absence d'activité nucléasique permet d'obtenir des fragments de taille différente puisque les ddNTPs ne sont pas excisés par le système de réparation. Pour vérifier le bon fonctionnement des réactions de séquence, un témoin, est séquencé en même temps que les individus étudiés. Le témoin utilisé est le plasmide PGEM (2
) d'un mélange réactionnel de séquençage, le Big Dye (PE Biosystems, ABI prism). Il contient les ddNTPs, les dNTPs, le MgCl2, le tampon et la Taq polymérase. L'absence d'activité nucléasique permet d'obtenir des fragments de taille différente puisque les ddNTPs ne sont pas excisés par le système de réparation. Pour vérifier le bon fonctionnement des réactions de séquence, un témoin, est séquencé en même temps que les individus étudiés. Le témoin utilisé est le plasmide PGEM (2 à 0.2
à 0.2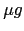 /
/ ) séquencé à l'aide du primer M13 (1
) séquencé à l'aide du primer M13 (1 à 0.8 pmole/
à 0.8 pmole/ ). Les conditions pour les réactions de séquence sont les suivantes : 96c 5 min, 96c 10 s, 55 ou 57c 5 s et 60c 4 min répété durant 25 cycles (20 cycles). Avant d'analyser les produits de la réaction de séquence sur le séquenceur, il est nécessaire de les purifier puis de les diluer. Cette purification se fait en déposant chacun de ces produits sur une colonne contenant du Sephadex G-50 Superfine (Amersham Pharmacia Biotech AB) et en les centrifugeant 2 minutes à 1100g. La dilution, permettant d'éviter un phénomène de saturation des pics lors de l'analyse, est effectuée en ajoutant 20
). Les conditions pour les réactions de séquence sont les suivantes : 96c 5 min, 96c 10 s, 55 ou 57c 5 s et 60c 4 min répété durant 25 cycles (20 cycles). Avant d'analyser les produits de la réaction de séquence sur le séquenceur, il est nécessaire de les purifier puis de les diluer. Cette purification se fait en déposant chacun de ces produits sur une colonne contenant du Sephadex G-50 Superfine (Amersham Pharmacia Biotech AB) et en les centrifugeant 2 minutes à 1100g. La dilution, permettant d'éviter un phénomène de saturation des pics lors de l'analyse, est effectuée en ajoutant 20 d'H2O pure au produit de séquence purifié. Les microplaques contenant les produits de séquences purifiés sont installées sur des séquenceurs à 96 capillaires, les 3700 ou 3730 DNA Analyser d'ABI-PRISM. Le traitement des résultats est réalisé par le programme GenalysWin [Takahashi et al., 2003]. Les résultats sont traduits en séquences sous forme d'une succession de pics des quatre couleurs représentant l'alternance des nucléotides au sein de la séquence (Figure 2.5Exemple d'une séquence visualisée par Genalys).Les séquences obtenues sont comparées à la séquence de référence du gène HLA-C, extraite d'une base de données (http://www.ebi.ac.uk/imgt/hla/).
d'H2O pure au produit de séquence purifié. Les microplaques contenant les produits de séquences purifiés sont installées sur des séquenceurs à 96 capillaires, les 3700 ou 3730 DNA Analyser d'ABI-PRISM. Le traitement des résultats est réalisé par le programme GenalysWin [Takahashi et al., 2003]. Les résultats sont traduits en séquences sous forme d'une succession de pics des quatre couleurs représentant l'alternance des nucléotides au sein de la séquence (Figure 2.5Exemple d'une séquence visualisée par Genalys).Les séquences obtenues sont comparées à la séquence de référence du gène HLA-C, extraite d'une base de données (http://www.ebi.ac.uk/imgt/hla/).
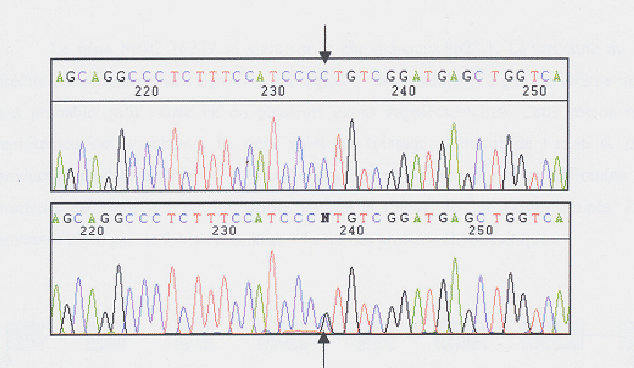
Figure 2.5:
Exemple d'une séquence visualisée par Genalys
Un polymorphisme est indiqué par une flèche. Un individu est homozygote pour la variation (en haut) et un autre individu est hétérozygote (en bas).
 |
Ce programme permet de présenter les résultats sous forme d'un tableau récapitulatif indiquant, pour chaque TagSNP d'intérêt, le génotype des 45 familles.




Next: Génotypage par la méthode
Up: Technique
Previous: Technique
Contents
anouar
2009-08-22
