Par la suite, deux analyses non paramétriques et multipoints (MLB et NPL) ont été réalisées, à l'aide des logiciels MLBGH et SIMWALK, sur le deuxième panel de 371 marqueurs sur les 46 familles (Criblage 2), suivies d'une analyse combinée sur l'ensemble des marqueurs génotypés lors des deux criblages sur ces mêmes familles (Criblage 1+2). Les résultats les plus significatifs de ces deux
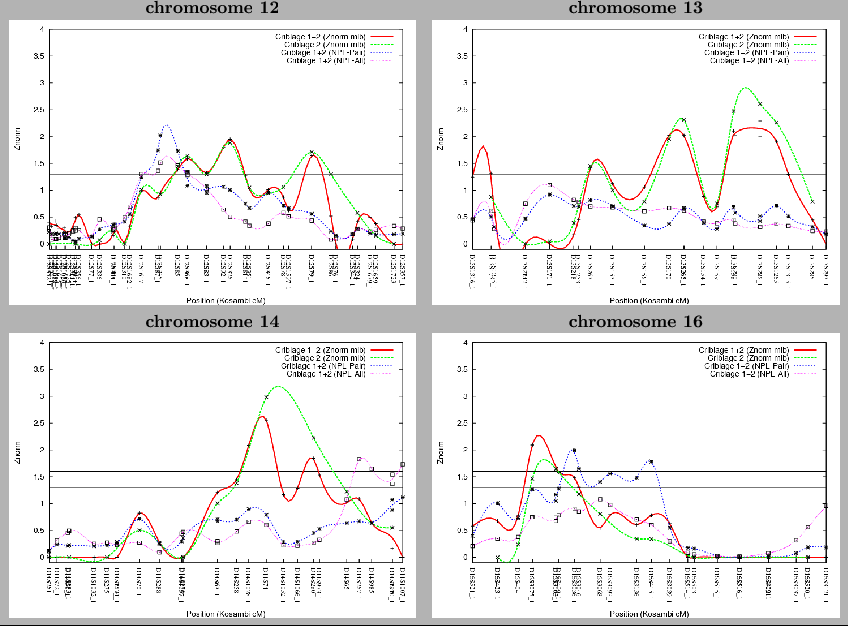
types d'analyses par les deux méthodes sont présentés dans la figure 3.1Résultats. Dans le cas de l'analyse non paramétrique par la méthode MLB, les résultats sont assez similaires entre l'étude du deuxième panel et celle combinée des deux panels. Ainsi, l'augmentation de la densité en marqueurs ne semble pas influencer l'analyse. En accord avec notre première étude, six régions 6p21, 10q, 13q, 14q, 16p et 20p13 sont retrouvées liées avec la maladie dans les 45 familles (P<0.05 ou Z![]() =1.6). Des liaisons de deux locus larges sur le chromosome 6, un en 6p23-12 (30-79 cM) et un autre en 6p12-q22 (80-130 cM) sont observées, avec des valeurs de P variant de 0.02 à 7.9x10
=1.6). Des liaisons de deux locus larges sur le chromosome 6, un en 6p23-12 (30-79 cM) et un autre en 6p12-q22 (80-130 cM) sont observées, avec des valeurs de P variant de 0.02 à 7.9x10![]() . La valeur de P la plus faible est obtenue pour un marqueur en 6p21 avec Z
. La valeur de P la plus faible est obtenue pour un marqueur en 6p21 avec Z![]() =3.78, proche du seuil théorique de significativité décrit par Lander et Kruglyak (1995) (P=2.2x10
=3.78, proche du seuil théorique de significativité décrit par Lander et Kruglyak (1995) (P=2.2x10![]() )[Lander and Kruglyak, 1995]. Pour les autres régions, les résultats restent similaires à ceux obtenus lors de notre première étude non paramétrique. Quatre loci potentiels sont suggérés lors des deux analyses (criblage 2 ou criblage 1+2) pour les régions 10q23-26 (Z
)[Lander and Kruglyak, 1995]. Pour les autres régions, les résultats restent similaires à ceux obtenus lors de notre première étude non paramétrique. Quatre loci potentiels sont suggérés lors des deux analyses (criblage 2 ou criblage 1+2) pour les régions 10q23-26 (Z![]() =2.5, P
=2.5, P![]() =0.006), 13q22-34 (Z
=0.006), 13q22-34 (Z![]() =2.6, P
=2.6, P![]() =0.005), 14q23-32 (Z
=0.005), 14q23-32 (Z![]() =3.0, P
=3.0, P![]() =0.001), 16p13-12 (Z
=0.001), 16p13-12 (Z![]() =2.1, P
=2.1, P![]() =0.02) et 20p13 (Z
=0.02) et 20p13 (Z![]() =2.2, P
=2.2, P![]() =0.01). Cinq nouveaux loci sur les chromosomes 1q, 7, 12q, 17q, 20p12, 20q13 et 22q sont également suggérés avec des valeurs de P
=0.01). Cinq nouveaux loci sur les chromosomes 1q, 7, 12q, 17q, 20p12, 20q13 et 22q sont également suggérés avec des valeurs de P![]() égales à 0.03 (Z
égales à 0.03 (Z![]() =1.9), 0.006 (Z
=1.9), 0.006 (Z![]() =2.5), 0.03 (Z
=2.5), 0.03 (Z![]() =1.9), 0.008 (Z
=1.9), 0.008 (Z![]() =2.4), 0.009 (Z
=2.4), 0.009 (Z![]() =2.3), 0.003 (Z
=2.3), 0.003 (Z![]() =2.7) et 0.002 (Z
=2.7) et 0.002 (Z![]() =2.9) respectivement.
=2.9) respectivement.
Dans le cas de l'analyse non paramétrique avec la méthode NPL, les résultats pour les deux statistiques (NPL![]() et NPL
et NPL![]() ) par SIMWALK2 sont assez similaires, excepté pour les régions des chromosomes 6, 16, 17 et 20, où de meilleurs résultats sont observés avec la statistique NPL
) par SIMWALK2 sont assez similaires, excepté pour les régions des chromosomes 6, 16, 17 et 20, où de meilleurs résultats sont observés avec la statistique NPL![]() . L'analyse du deuxième panel seul (371 marqueurs) sur les familles entières ne montre aucun résultat significatif (P<0.05 ou NPL=1.3) (données non montrées). La réduction de l'espacement entre marqueurs permet dans cette analyse multipoint d'augmenter la puissance de notre étude et d'identifier ou parfois de restreindre la localisation des loci d'intérêt, comparée à l'étude MLB.
. L'analyse du deuxième panel seul (371 marqueurs) sur les familles entières ne montre aucun résultat significatif (P<0.05 ou NPL=1.3) (données non montrées). La réduction de l'espacement entre marqueurs permet dans cette analyse multipoint d'augmenter la puissance de notre étude et d'identifier ou parfois de restreindre la localisation des loci d'intérêt, comparée à l'étude MLB.
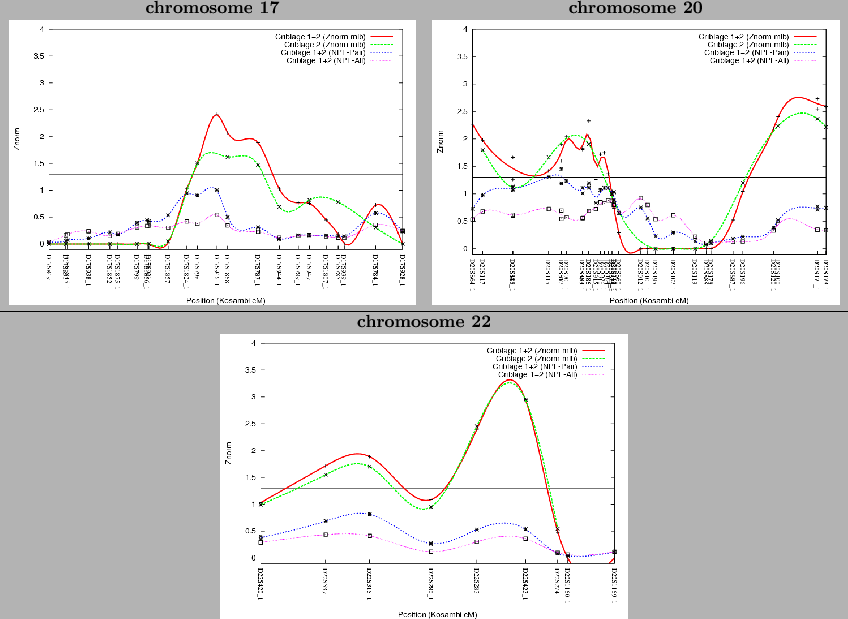
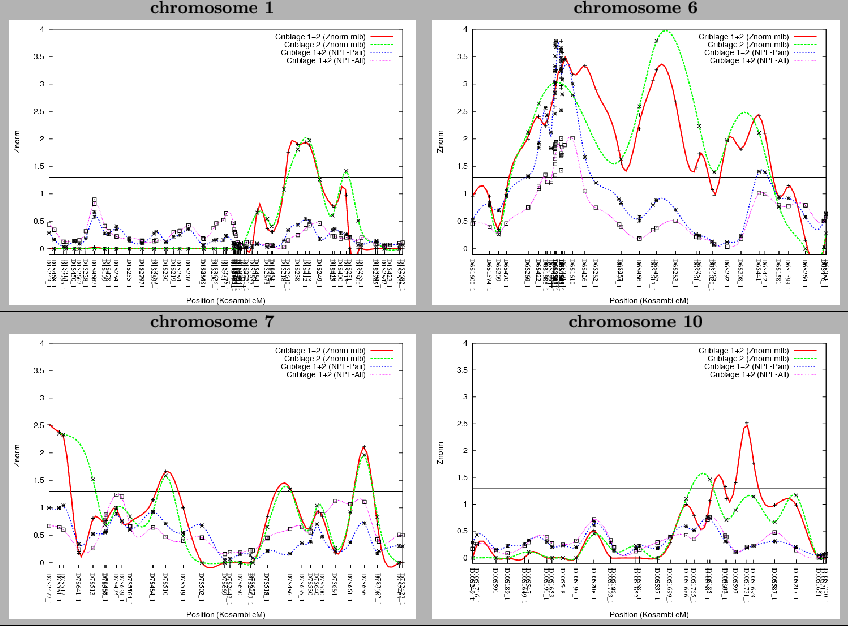
Lors de l'analyse globale des panels (679 marqueurs), la région 6p23-p21 est montrée fortement liée au psoriasis, particulièrement au niveau d'une région restreinte en 6p21 (NPL![]() = 3.78, P=1.66x10
= 3.78, P=1.66x10![]() ). Quatre autres régions sur les chromosomes 12, 14, 16 et 20 sont également retrouvées liées à la maladie mais de manière suggestive. Les régions correspondantes (p<0.05) sont néanmoins un peu différentes des analyses précédentes. Par exemple, les régions liées sont 12p12-q13 (NPL
). Quatre autres régions sur les chromosomes 12, 14, 16 et 20 sont également retrouvées liées à la maladie mais de manière suggestive. Les régions correspondantes (p<0.05) sont néanmoins un peu différentes des analyses précédentes. Par exemple, les régions liées sont 12p12-q13 (NPL![]() =2.02, P=0.01), 14q32 (NPL
=2.02, P=0.01), 14q32 (NPL![]() =1.73, P=0.02), 16p12-q12 (NPL
=1.73, P=0.02), 16p12-q12 (NPL![]() =1.99, P=0.01) et 20p12 (NPL
=1.99, P=0.01) et 20p12 (NPL![]() =1.45, P=0.04) au lieu de 12q13-q21, 14q24-32, 16p13-p12 et 20p13-p12. Aucune liaison significative n'est retrouvée pour les chromosomes 1, 7, 10, 13, 17 et 22.
=1.45, P=0.04) au lieu de 12q13-q21, 14q24-32, 16p13-p12 et 20p13-p12. Aucune liaison significative n'est retrouvée pour les chromosomes 1, 7, 10, 13, 17 et 22.
 |