



Next: ANALYSE D'ASSOCIATION: ETUDE DE
Up: Approfondissement de l'étude
Previous: Résultats
Contents
L'ensemble des analyses de liaison non paramétriques multipoints sur la totalité des familles confirme la liaison trouvée lors du premier criblage entre les régions 6p21, 14q, 16p, 20p et le psoriasis et révèle une nouvelle région en 12q. Les régions sur les chromosomes 1q25-32, 7p22-14, 10q23-26, 13q22-34, 17q11-23, 20q13 et 22q12-13 sont seulement montrées liées à la maladie lors de l'analyse MLB. Une liaison avec les loci 6p21, 7p14, 20p13 et 22q13 a également été trouvée lors des analyses paramétriques.
En raison du nombre théorique élevé de faux-positifs occasionnés lors des nombreux tests de criblages du génome, ces données doivent être interprétées avec précaution. Cependant, il est intéressant de noter que certains de ces loci soient révélés par deux méthodes différentes et/ou qu'ils aivaient été préalablement identifiés comme d'éventuels loci de prédisposition au psoriasis (en atteignant un seuil de P<0.05) dans au moins une autre étude indépendante.
Le pouvoir de détecter la liaison dépend de l'informativité des données, incluant celles obtenues par les structures des familles. Or, chaque méthode prend en compte différemment l'information familiale. Par exemple, la méthode MLB utilise l'information de tous les germains atteints prise globalement, ce qui est particulièrement intéressant dans le cas de grandes familles. L'application de cette méthode nécessite cependant la coupure des familles en familles nucléaires, ce qui n'est pas le cas avec le programme SIMWALK. L'analyse des familles entières permet d'augmenter la puissance de l'étude car l'analyse tient compte de la structure familiale pour estimer correctement le partage IBD. Ce pouvoir de détection peut cependant être diminué dans le cas d'une hétérogénéité génétique à l'intérieur des familles.
Sans détailler le rôle majeur du locus en 6p21 bien établi à l'heure actuelle, les régions 14q et 20p ont été déjà rapportées comme étant liées au psoriasis [Nair et al., 1997,Trembath et al., 1997,Veal et al., 2001,Karason et al., 2005,Liu et al., 2008].
Dans le cas du chromosome 14, une preuve suggestive de liaison a été initialement obtenue lors d'un criblage du génome de 158 familles anglaises (NPL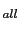 =2) au niveau de la région 14q32 pour un microsatellite, qui a également été retrouvée liée dans notre étude lors de l'analyse NPL [Veal et al., 2001]. Un autre criblage du génome de 259 patients de 98 familles islandaises, avec un psoriasis sensible aux traumatismes superficiels, a montré une liaison entre cette région 14q32 et le psoriasis (lod score=3.1)[Karason et al., 2005]. Dans une étude américaine récente d'association cas-témoins (810 versus 1256), réalisée sur l'ensemble du génome, une association avec un SNP dans le gène CLMN (calponin-like transmembrane domain protein) présent dans la région 14q32 (P=0.003) a été décrite [Liu et al., 2008].
=2) au niveau de la région 14q32 pour un microsatellite, qui a également été retrouvée liée dans notre étude lors de l'analyse NPL [Veal et al., 2001]. Un autre criblage du génome de 259 patients de 98 familles islandaises, avec un psoriasis sensible aux traumatismes superficiels, a montré une liaison entre cette région 14q32 et le psoriasis (lod score=3.1)[Karason et al., 2005]. Dans une étude américaine récente d'association cas-témoins (810 versus 1256), réalisée sur l'ensemble du génome, une association avec un SNP dans le gène CLMN (calponin-like transmembrane domain protein) présent dans la région 14q32 (P=0.003) a été décrite [Liu et al., 2008].
La région 20p était, dans notre précédente étude, la deuxième région liée le plus fortement au psoriasis après la région en 6p21. L'implication de ce locus a été déjà décrite dans deux autres études indépendantes: une première, sur 68 familles européennes, avec un marqueur en position 20p12 (P=0.0012, lod score=2.01) [Trembath et al., 1997]. Ceci a été confirmé par l'existence d'une liaison avec plusieurs marqueurs de la région 20p13-12 lors d'une autre étude réalisée sur 115 familles américaines et allemande s[Nair et al., 1997]. Le meilleur résultat est obtenu lors d'une analyse paramétrique sous le modèle récessif (Z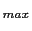 =2.6), ce qui est différent de notre étude paramétrique où la "liaison" est suggérée sous un modèle dominant [Nair et al., 1997]. Cependant, la meilleure preuve de liaison avec le chromosome 6 est observée lors de l'analyse paramétrique sous le modèle récessif des 115 familles (Z
=2.6), ce qui est différent de notre étude paramétrique où la "liaison" est suggérée sous un modèle dominant [Nair et al., 1997]. Cependant, la meilleure preuve de liaison avec le chromosome 6 est observée lors de l'analyse paramétrique sous le modèle récessif des 115 familles (Z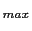 =2.6), ce qui est en accord avec notre étude.
La région 20p, particulièrement 20p13, a par conséquent été la première région étudiée par notre équipe à la suite du premier criblage des familles françaises, conduisant par la suite à la découverte d'une association avec un gène commun à d'autres maladies auto-immunes.
=2.6), ce qui est en accord avec notre étude.
La région 20p, particulièrement 20p13, a par conséquent été la première région étudiée par notre équipe à la suite du premier criblage des familles françaises, conduisant par la suite à la découverte d'une association avec un gène commun à d'autres maladies auto-immunes.
Aucune étude n'a été publiée pour une liaison significative entre le psoriasis et les régions 12q et 16p.
Parmi les autres régions suggérées lors de l'une de nos études, les régions 10q et 13q sont similaires aux régions identifiées lors d'une méta-analyse qui intègre les données de six criblages de génomes réalisés sur des populations différentes (422 familles caucasiennes et chinoises de structure différente) [Sagoo et al., 2004]. En plus de confirmer des régions déjà suggérées, cette méta-analyse a permis de trouver de nouveaux loci (dont 10q et 13q) où l'effet génétique était trop petit pour être détecté de manière significative par une seule de ces études. Dans le cas de la région 13q, le meilleur résultat est obtenu lors de l'étude de 61 familles chinoises (NPL=2.44, P=0.008) [Zhang et al., 2002]. Deux autres études, réalisées sur 710 familles caucasiennes et 79 familles islandaises, ont montrées une liaison entre la même région en 10q et le psoriasis (MLS=1.1 et lod score=3.1, respectivement) [Karason et al., 2005,Allen et al., 2003].
Il est également intéressant de noter que lors de cette méta-analyse, une deuxième région sur le chromosome 6 (66-99 cM), avec une liaison moins significative que le premier locus en 6p21, a été identifiée qui correspond à la région révélée par la méthode MLB, 6p12-q22 (80-130 cM).
La région en 22q12-13, montrée liée lors de nos analyses paramétriques et non paramétriques (avec MLB), a été également suggérée comme locus (NPL=2.2, P=0.02) lors d'une étude finlandaise de 31 familles [Asumalahti et al., 2003b]. De plus, cette étude montre un nouveau locus sur le chromosome 18p11 (NPL=3.58, P=0.003), qui contient le marqueur retrouvé lié dans notre étude paramétrique.
Cette région a aussi été identifiée dans une population anglaise (NPL=1.97). Cette dernière étude montre également une liaison avec une région commune entre plusieurs maladies auto-immunes en 7p15-q11 (NPL=2.1, P=0.0167), contenant le seul marqueur observé lié dans notre étude en 7p14 (D7S510) [Veal et al., 2001]. Dans notre analyse, deux des régions 1q25-32 et 17q11-23 sont proches des régions de prédisposition du psoriasis connues PSORS4 (1q21) et PSORS2 (17q25) [Nair et al., 1997,Capon et al., 1999b]. Les régions 7p22, 20q13 ne sont a priori pas décrites dans une autre étude.
Ces régions, caractérisées comme contenant un ou des gènes potentiellement impliqués dans la maladie, sont seulement répliquées dans une ou deux études et ces études ne montrent, dans la plupart des cas, qu'une liaison suggestive avec une valeur de P non corrigée proche du seuil de 5%. Le seul locus, retrouvé dans la majorité des études, est le locus HLA et, dans notre étude il est le seul locus à être fortement lié au psoriasis avec une valeur de P <0.001 dans l'ensemble de nos analyses.
Pour l'identification de régions d'intérêt, une preuve de liaison obtenue dans de nombreuses études peut être plus éloquente qu'une seule liaison significative non répliquée.
Pour les autres loci, l'absence de réplication dans d'autres études peut indiquer que ces loci peuvent être des faux-positifs (dus aux multiples tests), être spécifiques à une population ou être la conséquence d'un manque de puissance pour les détecter. La puissance d'une étude est dépendante entre autres de la taille de l'échantillon étudié, de la contribution du gène à identifier (effet majeur ou mineur), de la présence d'une hétérogénéité (phénotypique ou génétique), de l'informativité des marqueurs. Dans le cas du psoriasis, l'hétérogénéité phénotypique ou génétique au sein d'un échantillon peut être importante et peu d'études ont été réalisées sur de grands échantillons, pouvant limiter l'identification de gènes à effet mineur. L'hétérogénéité phénotypique est théoriquement réduite dans notre étude, concernant 45 grandes familles avec une grande majorité des malades (environ 96%) atteints de psoriasis en plaques. Néanmoins, en raison de la fréquence élevée de la maladie en France (4%), une hétérogénéité génétique au sein d'une même famille peut exister, par suite d'un mariage entre un individu atteint de la famille étudiée et un conjoint lui-même malade. Ce dernier cas a d'ailleurs été noté dans certaines de nos familles. De plus, la plupart des études discutées précédemment ne présentent que les résultats d'un criblage à maille large du génome. Peu de ces régions ont été étudiées de manière plus précises, en augmentant par exemple la densité des marqueurs. Comme observé lors d'une de nos études non-paramétriques, l'analyse de marqueurs supplémentaires pour réaliser un criblage plus fin peut permettre une meilleure exploitation des données, surtout dans le cas d'une analyse multipoint.
Ainsi, l'ensemble des ces résultats doit nécessairement être répliqué par plusieurs études et, si possible dans les meilleures conditions, telles que l'étude d'échantillons de plus grande taille.




Next: ANALYSE D'ASSOCIATION: ETUDE DE
Up: Approfondissement de l'étude
Previous: Résultats
Contents
anouar
2009-08-22