



Next: Contrôle qualité général
Up: Génotypage par la méthode
Previous: Principe détaillé
Contents
La plupart du génotypage des SNPs étudiés a été réalisée en utilisant la technique SNPlex (Applied Biosystems, Foster City, CA, USA). Deux panels ont été ainsi effectués pour le génotypage de 93 SNPs. Le génotypage des SNPs des gènes FLG, CYLD, SLC9A3R1, NAT9, raptor, IL12RB1, TGM3 et TGM6, ainsi que celui d'une vingtaine de SNPs non possibles par la méthode SNPlex ont été réalisés par la technique Taqman
(Applied Biosystems, Foster City, CA, USA). Deux panels ont été ainsi effectués pour le génotypage de 93 SNPs. Le génotypage des SNPs des gènes FLG, CYLD, SLC9A3R1, NAT9, raptor, IL12RB1, TGM3 et TGM6, ainsi que celui d'une vingtaine de SNPs non possibles par la méthode SNPlex ont été réalisés par la technique Taqman .
.
Le choix de la combinaison optimale des 48 SNPs, pour être dans les meilleures conditions pour la réaction multiplex, ainsi que l'ensemble des sondes SNPlex spécifiques des SNPs des deux panels (93 SNPs) ont été déterminés et obtenus par le service en ligne "online SNP selection" de Applied Biosystems (http://myscience.appliedbiosystems.com). Dans cette technique, le génotypage est réalisé à partir de 50 ng d'ADN fragmenté et séché dans des plaques 384, en suivant les instructions du fournisseur Applied Biosystems. Cette technique SNPlex se fait en plusieurs étapes, résumées sur la figure 2.8Résumé des différentes étapes de la méthode SNPlex:
- La première étape est l'étape de préparation des ADNs (mise en plaques 384, fragmentation, dilution). Pour chaque plaque de 96 puits de départ, nous utilisons deux ADNs CEPH comme témoins positifs, un témoin négatif
Figure 2.8:
Résumé des différentes étapes de la méthode SNPlex
(D'après le site: http://www.rockefeller.edu/genomics/pdf/Applied_Biosystems_SNPlex_Genotyping_Manual.pdf)
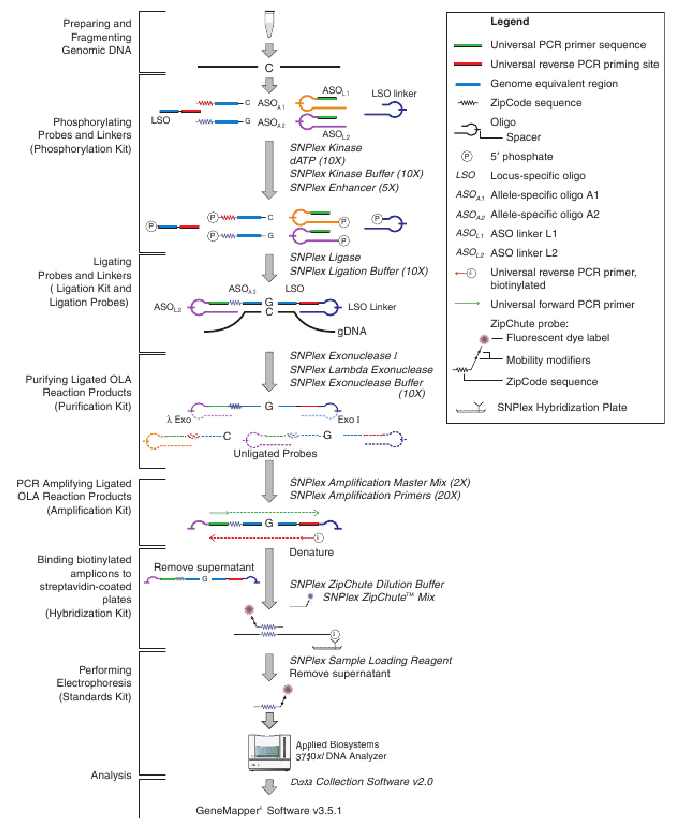 |
(l'eau) et deux puits vides (G12, H12) qui seront utilisés lors du SNPlex pour les marqueurs de calibration des sondes "ZipChute".
- La deuxième étape consiste à préparer les différentes sondes (96"ASO"/48 "LSO") et les linkers (96 "ASO"/ 1 "LSO") en les phosphorylisant en 5' pour l'étape dite réaction "OLA".
- La troisième étape est de rassembler l'ensemble des sondes activées et l'ADN et de permettre la liaison ciblée des sondes sur celui-ci par une ligase. C'est la réaction "OLA".
- La quatrième étape est de purifier les échantillons en enlevant les produits non liés lors de l'étape de ligation en utilisant deux exonucléases (Exonucléase I et Lambda).
- La cinquième étape est d'amplifier, de façon globale grâce à des amorces universelles, l'ensemble des produits obtenus et purifiés lors de la réaction "OLA". Cette amplification par PCR est rendue possible, par la présence sur les linkers, des deux "ASO" et de "LSO" de séquences complémentaires aux amorces sens et anti-sens universelles, respectivement, utilisées pour amplifier tous les produits obtenus lors de la réaction OLA. L'amorce anti-sens universelle a une particularité, elle est biotinylée, entrainant ainsi une forte affinité pour la streptavidine. Les produits obtenus lors de cette PCR seront donc des amplicons double-brins avec un brin biotinylé.
- La dernière étape est nécessaire pour obtenir les sondes ZipChute dans la plaque à charger. Après fixation des fragments double-brins biotinylés à la streptavidine dans les puits d'une plaque dite "d'hybridation", un lavage aide à enlever le matériel non fixé. Une dénaturation permet de garder un seul brin, celui biotinylé et fixé à la streptavidine. Ceci favorise ainsi la fixation des sondes "ZipChutes" fluorescentes sur la partie "ZipCode" du fragment simple brin biotinylé par complémentarité. Le relargage des sondes "ZipChutes" est ensuite réalisé dans une solution dite de chargement, utilisée pour établir une courbe de calibration de la taille grâce à 11 marqueurs fluorescents afin de pouvoir identifier les différentes sondes "ZipChute". Elles sont enfin chargées dans la machine ABI PRISM® 3730XL DNA Analyzer, puis traitées et analysées, grâce au programme d'analyse "GeneMapper", pour avoir le génotype des 48 SNPs par individu (Applied Biosystems, Foster City, CA, USA).
Tous les étapes possèdent une étape de lavage des canules ainsi que l'utilisation de différentes solutions (l'eau milliQ, UNG (uracyl-N-Glycosylase)) dans différentes salles (pré ou post-PCR) afin d'éviter toute contamination.




Next: Contrôle qualité général
Up: Génotypage par la méthode
Previous: Principe détaillé
Contents
anouar
2009-08-22