



Next: Principe
Up: Contexte
Previous: Contexte sur le locus
Contents
Premier criblage
Après la découverte d'un locus de prédisposition au psoriasis sur le chromosome 20p13 dans notre étude de liaison, nous avons réalisé une étude de cartographie fine de ce large locus de 17Mb sur nos 45 familles afin d'identifier le gène de prédisposition parmi les nombreux gènes de la région. Pour réaliser un premier criblage de l'intervalle avec un nombre limité de SNPs, les SNPs ont été sélectionnés en fonction de plusieurs critères : 1- ils sont identifiés dans la population d'origine européenne CEU du projet HapMap (30 trios résidents des États-Unis originaires de l'Europe du Nord et de l'Ouest, receuillis par le Centre d'Étude du Polymorphisme Humain (CEPH) (([Consortium, 2003]),
 ), 2- ils doivent avoir une fréquence de l'allèle mineur supérieur (MAF) à 20% dans cette population d'origine européenne, 3- ils sont présents dans ou proches des gènes connus (+10kb en amont et en aval des gènes connus), 4- ils ont des séquences flanquantes non ambigües. Au total, 85 SNPs, décrits dans le premier tableau supplémentaire de l'article, sont sélectionnés pour le premier criblage et ne sont pas en DL entre eux (1 SNP/137 kb en moyenne).
), 2- ils doivent avoir une fréquence de l'allèle mineur supérieur (MAF) à 20% dans cette population d'origine européenne, 3- ils sont présents dans ou proches des gènes connus (+10kb en amont et en aval des gènes connus), 4- ils ont des séquences flanquantes non ambigües. Au total, 85 SNPs, décrits dans le premier tableau supplémentaire de l'article, sont sélectionnés pour le premier criblage et ne sont pas en DL entre eux (1 SNP/137 kb en moyenne).
Deuxième criblage
Une étude plus approfondie de trois régions distinctes (locus DEFB, locus ADAM33 et locus AK125948), dont proviennent 4 SNPs montrés associés dans le premier criblage, a été réalisée en majorité par une approche "TagSNPs". Cette stratégie a été utilisée lorsque la couverture du gène en SNPs est suffisante dans la base de données Hapmap car elle se base sur le DL au sein du gène pour choisir le minimum suffisant de SNPs spécifiques, nommés les "TagSNPs", pour représenter
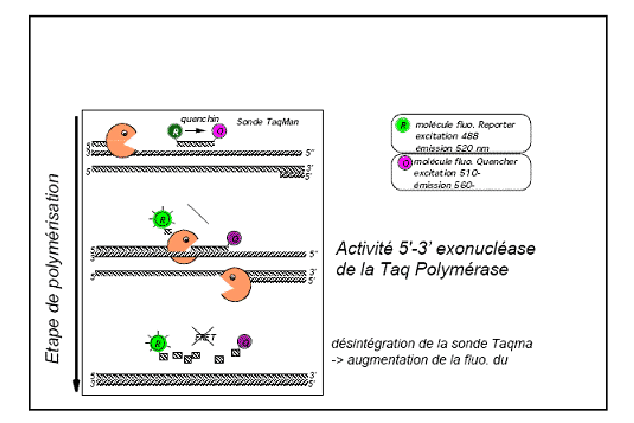
Figure 2.6:
Représentation des étapes de polymérisation dans la technique TAQMAN
(D'après le site: http://www1.montpellier.inra.fr/biochimie/td/UB/TD/VMJ-taqman.pdf)
 |
toute la diversité haplotypique commune du gène [Consortium, 2005,de Bakker et al., 2005]. Ce fut le cas pour les gènes SIGLEC1 (17 TagSNPs sélectionnés) et AK125945 (5 TagSNPs sélectionnés). Pour ces deux gènes, deux SNPs communs absents dans la base de données HapMap (rs1018493 dans l'exon 6 de SIGLEC1 et rs1060236, dans la région UTR de AK125945) ont été rajoutés dans l'analyse des 45 familles.
Lorsque la densité en SNPs connus par HapMap n'est pas suffisante au sein du gène candidat, ce qui est le cas pour le gène ADAM33, nous avons augmenté le nombre de SNPs à analyser en intégrant les SNPs validés dans la base de données dbSNP ou dans la littérature. Le gène ADAM33 a été préalablement séquencé lors de plusieurs études et certains SNPs décrits n'existaient pas dans la base HapMap [Van Eerdewegh et al., 2002,Chae et al., 2003]. Ainsi, en plus des 8 TagSNPs de HapMap, 15 autres marqueurs ont été pris en compte lors de cette analyse. Parmi ces SNPs, 9 SNPs (rs3918392, rs511898, rs612709, rs3918396, rs44707, rs574174, rs628977, rs543749 et rs2787094) ont été montrés associés avec l'asthme [Van Eerdewegh et al., 2002]. Pour GFRA4, peu de SNPs ont été identifiés et sélectionnés (4 SNPs), bien que la séquence codante entière du gène ait été re-séquencée [Cebrian et al., 2005]. En raison des faibles données dans les bases de données sur les gènes DEFB125, DEFB126, DEFB127, DEFB128, DEFB129 et DEFB32, nous avons reséquencé la région codante et les jonctions exon/intron de ces gènes chez 58 témoins caucasiens indépendants. L'identification des TagSNPs au sein de ces gènes a été réalisée à partir du programme tagsnps [Stram, 2004].
Au total, 63 SNPs supplémentaires ont été analysés pour l'étude fine de la région.




Next: Principe
Up: Contexte
Previous: Contexte sur le locus
Contents
anouar
2009-08-22
