



Next: Principe
Up: Génotypage par la méthode
Previous: Génotypage par la méthode
Contents
La stratégie "gènes candidats" consiste à étudier des gènes dont la fonction pourrait être impliqué dans l'étiologie de la maladie. Les gènes candidats évidents pour une maladie auto-immune et inflammatoire telle que le psoriasis sont les gènes impliqués dans la régulation du système immunitaire et de l'inflammation. De plus, le psoriasis étant une maladie de peau, cela inclut un rôle particulier des kératinocytes. Les autres candidats potentiels sont les gènes intervenant dans la survie, la prolifération et la différenciation des kératinocytes mais également intervenant de manière générale dans la structure de la peau.
L'observation d'une physiopathologie commune entre diverses maladies et celle d'une forte agrégation des maladies dans certaines familles suggère fortement l'existence de mécanismes communs, impliquant entre autres des facteurs génétiques. Ceci fut d'ailleurs constaté avec l'identification de loci communs à plusieurs maladies auto-immunes et inflammatoires. Ainsi, 22 gènes (PTPN22, CARD15, CYLD, FLG, IL1R1, IL1A, IL1B, IL1RN, SLC12A8, SLC22A4, SLC22A5, NAT9, SLC9A3R1, raptor, IL12RB1, TGM1, TGM3, TGM5, TGM6, JUN, JUNB et STAT3) ont été sélectionnés en fonction de leur rôle dans les processus métaboliques de l'immunité ou de la peau ainsi qu'en fonction de l'existence de leur association avec d'autres maladies auto-immunes ou d'autres maladies de la peau incluant le psoriasis.
L'implication d'un gène commun dans différentes maladies ne signifie pas l'existence d'un même variant causal; une approche systémique "Tagging SNP" a été entreprise pour tester tous les gènes éventuellement en cause. Comme précédemment décrit, cette approche utilise l'information d'un minimum de SNPs qui sont représentatifs de la diversité allélique dans la région du gène, nommé "TagSNPs". Le choix de ces SNPs "représentatifs" dépend du niveau de déséquilibre de liaison (DL) entre les polymorphismes de la région étudiée (ou "structure DL"). Pour sélectionner les "TagSNPs", nous avons procédé de différentes manières qui ont dépendu de la disponibilité de données génétiques sur les SNPs présents dans les différents gènes et de la procédure technique employée à ce moment-là. Pour la plupart des gènes, les SNPs sont identifiés dans la base de données HapMap et choisis selon les critères suivants :1- ils doivent être "validés" dans la base HapMap ([Consortium, 2005]) -2 ils doivent être des polymorphismes bi-alléliques dans la population caucasienne (CEU) répertoriés dans cette base (précédemment décrite) et ils doivent avoir aussi des séquences flanquantes non ambigües pour des raisons techniques dues aux méthodes de génotypage employées (Taqman et SNPlex),-3 ils doivent avoir une fréquence de l'allèle mineur (MAF) supérieure ou égale à 5% dans la population caucasienne (CEU). Le choix de ce seuil reste indicatif. Afin d'identifier une association entre le SNP et la maladie, il est nécessaire de choisir des SNPs assez communs dans la population générale pour obtenir une puissance d'analyse suffisante. Si le SNP est trop rare, la plupart des individus étudiés seront homozygotes pour ce marqueur, et par conséquent, il sera difficile d'avoir suffisamment de familles nucléaires informatives pour une étude de type TDT qui repose sur la transmission d'allèles d'un parent hétérozygote à son enfant atteint, -4 ils doivent être présents dans le gène candidat lui-même (c'est le cas pour les gènes : PTPN22, IL1R1, IL1A, IL1B, IL1RN, SLC12A8, SLC22A5, TGM1, TGM3, TGM5, TGM6 et CARD15) ou présents autour et dans le gène (par exemple, +10 kb en amont et aval des gènes JUN, JUNB et STAT3, ou + 20 kb pour les gènes CYLD et FLG).
Après cette première sélection, nous avons limité au minimum le nombre de SNPs à génotyper en tenant compte, par gène, du déséquilibre de liaison entre les SNPs pris deux à deux. Lorsque la couverture du gène en SNPs est suffisante dans la base de données Hapmap, nous avons sélectionné, par le programme Haploview, les SNPs représentatifs de la variation allélique commune (MAF 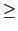 0.05) en se basant sur la présence d'un fort DL entre deux variants pour un
0.05) en se basant sur la présence d'un fort DL entre deux variants pour un 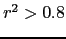 [de Bakker et al., 2005]. Les données HapMap sont disponibles via http://www.hapmap.org (provenant de Janvier 2007 pour FLG et CYLD, Octobre 2005 pour JUN, JUNB et Stat3 et enfin, Juin 2005 pour les autres gènes).
[de Bakker et al., 2005]. Les données HapMap sont disponibles via http://www.hapmap.org (provenant de Janvier 2007 pour FLG et CYLD, Octobre 2005 pour JUN, JUNB et Stat3 et enfin, Juin 2005 pour les autres gènes).
Dans le cas d'une couverture insuffisante du gène, la densité de marqueur est augmentée en ajoutant dans l'analyse des SNPs validés dans les populations caucasiennes provenant d'autres bases de données comme dbSNP
(http://www.ncbi.nlm.nih.gov/SNP/), Ensembl (http://www.ensembl.org/) et Perlegen (http://genome.perlegen.com/).
En parallèle, nous avons incorporé les SNPs montrés associés avec des maladies auto-immunes ou des maladies de la peau. C'est le cas de PTPN22 où un SNP fonctionnel (rs2476601) a été trouvé associé avec différentes maladies auto-immunes comme le diabète de type 1, la polyarthrite rhumatoïde, le lupus érythémateux systémique (méta-analyse [Lee et al., 2007]). Pour le gène de la filagrine (FLG), trois mutations à type de "perte de fonction" R501X, 2282del4, et 3702delG sont associées avec la dermatite atopique, l'asthme associé avec l'eczéma et l'ichtyose vulgaire dans plusieurs populations européennes [Palmer et al., 2006,Palmer et al., 2007,Sandilands et al., 2006,Smith et al., 2006]. Pour le gène IL1A, le SNP rs1800587 confère une prédisposition à la spondylarthrite ankylosante [Wu and Gu, 2007]. Pour le gène SLC12A8, 5 SNPs (rs651630, rs9831295, rs9813946, rs28986275 (ss35527511), rs28986277 (ss35527513)) sont montrés fortement associés avec le psoriasis dans une population allemande [Huffmeier et al., 2005a]. Pour le gène CARD15, 3 SNPs (SNP 8 (rs2066844), SNP12 (rs2066845), SNP13 (rs2066847)) prédisposent à la maladie de Crohn et également (pour un) à l'arthrite psoriasique (PsA) [Hugot et al., 2001,Ogura et al., 2001,Rahman et al., 2003]. Trois autres SNPs ont été associés avec une augmentation du risque de développer l'asthme (rs3135500) ou avec une diminution du risque de développer l'asthme (rs1077861) ou la rhino-conjonctivite allergique (rs2076752) [Weidinger et al., 2005].
Pour 4 des gènes candidats (SLC22A4, SLC9A3R1/NAT9, raptor, IL12RB1), nous avons répliqué les SNPs étudiés dans des études d'association réalisées sur des maladies auto-immunes. Pour SLC22A4, 5 SNPs (individuellement (rs3792876) et sous forme d'un haplotype (rs3763112, rs1007602, rs2073838, rs2269822)) sont associés avec la polyarthrite rhumatoïde dans une population japonaise [Tokuhiro et al., 2003]. En plus, deux SNPs (rs1050152, rs2631367), présents à l'intérieur du gène SLC22A4 et l'autre dans le promoteur du gène SLC22A5 ont été intégrés dans cette étude car ils forment un haplotype associé avec la maladie de Crohn [Peltekova et al., 2004]. Pour la région SLC9A3R1/NAT9, 5 SNPs (rs7420, rs734232, rs895691, rs12797 et rs2305214), et trois autres SNPs (rs1564864, rs2019154, rs869190) présents au niveau du gène RAPTOR, ont été sélectionnés pour leurs associations avec le psoriasis [Helms et al., 2003]. Pour le gène IL12RB1, nous avons intégré directement les 8 TagSNPs (rs404733, rs383483, rs17882636, rs375947, rs11086087, rs2305743, rs436857, rs393548) décrits dont deux sont associés dans une précédente étude de prédisposition à la dermatite atopique [Takahashi et al., 2005].
Les provenances des 153 SNPs choisis pour être génotypés dans le Lot I sont indiquées dans les tableaux du chapitre 3 (Analyse d'association: Etude de gènes candidats).




Next: Principe
Up: Génotypage par la méthode
Previous: Génotypage par la méthode
Contents
anouar
2009-08-22