Mais D est également estimé par:
|
Mais D est également estimé par: |
|
où et |
 |
population. Ainsi, contrairement au cas où ![]() , deux SNPs ayant un
, deux SNPs ayant un ![]() de 1 sont totalement équivalents et l'un peut être utilisé comme substitut de l'autre (l'information contenue est équivalente). Ceci est dû à la présence de seulement deux des génotypes possibles. En pratique, un
de 1 sont totalement équivalents et l'un peut être utilisé comme substitut de l'autre (l'information contenue est équivalente). Ceci est dû à la présence de seulement deux des génotypes possibles. En pratique, un ![]() de 0,8 est habituellement choisi dans les études d'association pour décrire deux SNPs équivalents.
de 0,8 est habituellement choisi dans les études d'association pour décrire deux SNPs équivalents.
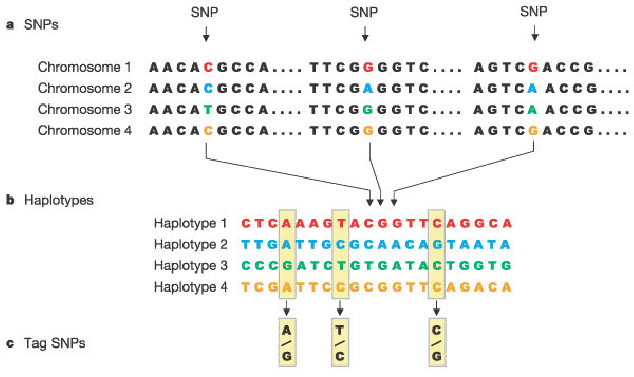
L'étude du DL au sein du génome montre que le DL peut parfois s'étendre sur plusieurs centaines de kilobases. Le génome est d'ailleurs structuré en fonction du DL, résultant d'une différence du taux de recombinaison le long du génome. Cette structuration en blocs de DL peut aussi résulter de phénomènes dépendants de la population étudiée tels que la dérive génétique (fixation d'un allèle de manière aléatoire au sein de la population), la croissance et la stratification de la population, la sélection naturelle et l'apparition de mutations. Le génome est donc organisé en blocs de régions transmises de manière intacte au cours des générations. Ces blocs dans lesquels il existe un fort DL et par conséquent, une faible diversité haplotypique (soit les différentes combinaisons possibles des allèles de chaque SNP) sont appelés des blocs de DL (ou blocs haplotypiques). Ainsi, par l'existence de ce fort DL, de nombreux polymorphismes donnent la même information sur la variation génétique au sein du bloc car ils sont souvent hérités ensemble. Par conséquent, seulement quelques polymorphismes seront nécessaires à étudier car ils seront représentatifs de l'ensemble des haplotypes existants dans la population, c'est-à-dire de l'ensemble de la variabilité génétique du bloc. Ils sont nommés "TagSNPs" (Figure 2.3Approche "TagSNPs" se fait en trois étapes).
La caractérisation des SNPs, en particulier des TagSNPs, a été l'objectif de différents projets d'analyse du génome dont HapMap (http://www.hapmap.org/) afin de faciliter les études d'association [Consortium, 2003]. Ce projet international a permis de cataloguer les variations génétiques (nature, position et fréquence selon les populations) les plus fréquentes chez l'Humain, en analysant l'ADN de 4 populations d'origine africaine, asiatique (japonaise et chinoise) et européenne [Consortium, 2003]. Cette connaissance permet l'identification de la plupart des haplotypes communs à la population mondiale, variables en fréquence selon les populations. Ainsi, l'étude des blocs DL en fonction des populations permet de caractériser les identificateurs uniques d'un haplotype, les TagSNPs, spécifique d'une population [Consortium, 2005]. Cela est très utile pour des études d'association systématiques.