



Next: Analyse paramétrique
Up: Analyses paramétriques
Previous: Analyses paramétriques
Contents
L'analyse paramétrique, qui teste la vraisemblance de la liaison de la maladie à des marqueurs dans des familles selon différentes valeurs du taux de recombinaison 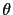 , visualisé par le lod score, dépend de différents paramètres: le modèle génétique, les caractéristiques de la maladie (autosomique dominante, récessive, fréquence du gène, pénétrance..) et les caractéristiques des familles étudiées (nombre de sujets atteints, consanguinité..). Comme exposé dans l'introduction, dans le cas des maladies à hérédité complexe, ces paramètres sont souvent difficiles à estimer et peuvent entraîner de nombreux faux-positifs. Cependant, dans notre recherche, il pouvait être intéressant de faire une étude préalable par une analyse paramétrique car nos échantillons sont des grandes familles avec plusieurs générations et avec une transmission apparemment autosomique dominante du psoriasis. Avant de réaliser le génotypage, il est important d'estimer la probabilité de détecter une liaison dans des conditions définies (le modèle génétique, les caractéristiques de la maladie). La puissance théorique d'analyse paramétrique bi-point des familles est estimée par le programme SLINK (option MSIM) qui calcule, par simulation, la probabilité d'obtenir un lod score maximal à partir des différentes données choisies au préalable [Weeks et al., 1990]. Pour tester nos familles, les hypothèses choisies sont les suivantes : mode de transmission autosomique dominant avec une pénétrance incomplète (80%), absence d'hétérogénéité génétique, fréquence allélique de la maladie de 1% et analyse d'un marqueur avec quatre allèles équi-fréquents. Après une simulation de 500 réplications, le lod score maximum moyen estimé à
, visualisé par le lod score, dépend de différents paramètres: le modèle génétique, les caractéristiques de la maladie (autosomique dominante, récessive, fréquence du gène, pénétrance..) et les caractéristiques des familles étudiées (nombre de sujets atteints, consanguinité..). Comme exposé dans l'introduction, dans le cas des maladies à hérédité complexe, ces paramètres sont souvent difficiles à estimer et peuvent entraîner de nombreux faux-positifs. Cependant, dans notre recherche, il pouvait être intéressant de faire une étude préalable par une analyse paramétrique car nos échantillons sont des grandes familles avec plusieurs générations et avec une transmission apparemment autosomique dominante du psoriasis. Avant de réaliser le génotypage, il est important d'estimer la probabilité de détecter une liaison dans des conditions définies (le modèle génétique, les caractéristiques de la maladie). La puissance théorique d'analyse paramétrique bi-point des familles est estimée par le programme SLINK (option MSIM) qui calcule, par simulation, la probabilité d'obtenir un lod score maximal à partir des différentes données choisies au préalable [Weeks et al., 1990]. Pour tester nos familles, les hypothèses choisies sont les suivantes : mode de transmission autosomique dominant avec une pénétrance incomplète (80%), absence d'hétérogénéité génétique, fréquence allélique de la maladie de 1% et analyse d'un marqueur avec quatre allèles équi-fréquents. Après une simulation de 500 réplications, le lod score maximum moyen estimé à 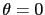 est supérieur à 3 dans 42% (19/45) des familles. Il est entre 2 et 3 dans 22% (10/45) des familles,
est supérieur à 3 dans 42% (19/45) des familles. Il est entre 2 et 3 dans 22% (10/45) des familles, 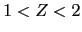 dans 25% (11/45) et <1 pour le reste de familles (11%, 5/45). Le lod score moyen pour toutes les familles est de 3.2 (l'écart type: 1.19) avec comme valeur la plus haute 7.64 et la plus basse 0.30. Or, le seuil de décision pour que l'hypothèse d'indépendance soit rejetée est de
dans 25% (11/45) et <1 pour le reste de familles (11%, 5/45). Le lod score moyen pour toutes les familles est de 3.2 (l'écart type: 1.19) avec comme valeur la plus haute 7.64 et la plus basse 0.30. Or, le seuil de décision pour que l'hypothèse d'indépendance soit rejetée est de 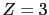 . Même si une analyse paramétrique est peu adaptée dans le cas d'une "maladie complexe" pour identifier des régions de prédisposition, cette analyse a été réalisée sous deux modèles, en raison de la puissance théorique élevée de certaines de nos grandes familles.
. Même si une analyse paramétrique est peu adaptée dans le cas d'une "maladie complexe" pour identifier des régions de prédisposition, cette analyse a été réalisée sous deux modèles, en raison de la puissance théorique élevée de certaines de nos grandes familles.




Next: Analyse paramétrique
Up: Analyses paramétriques
Previous: Analyses paramétriques
Contents
anouar
2009-08-22